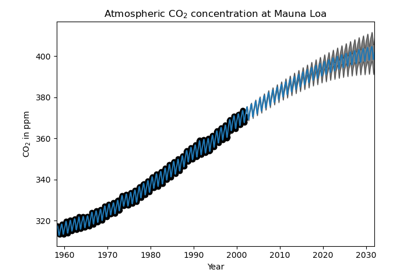
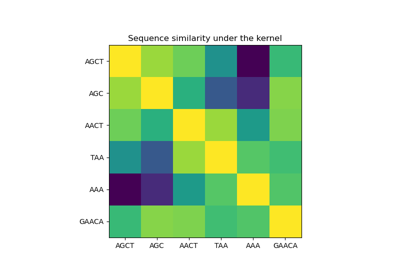
sklearn.gaussian_process.GaussianProcessRegressor¶
- class sklearn.gaussian_process.GaussianProcessRegressor¶
Regresión de procesos gaussianos (Gaussian process regression, GPR).
La implementación se basa en el algoritmo 2.1 de Gaussian Processes for Machine Learning (GPML) de Rasmussen y Williams.
Además de la API del estimador estándar de scikit-learn, GaussianProcessRegressor:
permite la predicción sin ajuste previo (basado en el previo GP)
proporciona un método adicional
sample_y(X), que evalúa las muestras extraídas del GPR (a priori o a posteriori) en entradas específicasexpone un método
log_marginal_likelihood(theta), que puede ser usado externamente para otras maneras de seleccionar hiperparámetros, por ejemplo, a través de la cadena de Markov Monte Carlo.
Más información en el Manual de usuario.
Nuevo en la versión 0.18.
- Parámetros
- kernelinstancia del núcleo, default=None
El núcleo que especifica la función de covarianza de la GP. Si se pasa None, el núcleo
ConstantKernel(1.0, constant_value_bounds="fixed" * RBF(1.0, length_scale_bounds="fixed")se utiliza por defecto. Ten en cuenta que los hiperparámetros del núcleo se optimizan durante el ajuste a menos que los límites estén marcados como «fijos».- alphafloat o ndarray de forma (n_samples,), default=1e-10
Valor añadido a la diagonal de la matriz del núcleo durante el ajuste. Esto puede evitar un posible problema numérico durante el ajuste, garantizando que los valores calculados formen una matriz positiva definida. También puede interpretarse como la varianza del ruido de medición gaussiano adicional en las observaciones de entrenamiento. Ten en cuenta que esto es diferente de utilizar un
WhiteKernel. Si se pasa un arreglo, debe tener el mismo número de entradas que los datos utilizados para el ajuste y se utiliza como nivel de ruido dependiente del punto de datos. Permitir especificar el nivel de ruido directamente como un parámetro es principalmente por conveniencia y por consistencia con Ridge.- optimizer«fmin_l_bfgs_b» o invocable, default=»fmin_l_bfgs_b»
Puede ser uno de los optimizadores soportados internamente para optimizar los parámetros del núcleo, especificado por una cadena, o un optimizador definido externamente pasado como invocable. Si se pasa un invocable, debe tener la firma (signature):
def optimizer(obj_func, initial_theta, bounds): # * 'obj_func' is the objective function to be minimized, which # takes the hyperparameters theta as parameter and an # optional flag eval_gradient, which determines if the # gradient is returned additionally to the function value # * 'initial_theta': the initial value for theta, which can be # used by local optimizers # * 'bounds': the bounds on the values of theta .... # Returned are the best found hyperparameters theta and # the corresponding value of the target function. return theta_opt, func_min
Por defecto, se utiliza el algoritmo “L-BFGS-B” de scipy.optimize.minimize. Si se pasa None, los parámetros del núcleo se mantienen fijos. Los optimizadores internos disponibles son:
'fmin_l_bfgs_b'- n_restarts_optimizerint, default=0
El número de reinicios del optimizador para encontrar los parámetros del núcleo que maximizan la verosimilitud logarítmica marginal. La primera ejecución del optimizador se realiza a partir de los parámetros iniciales del núcleo, los restantes (si los hay) a partir de los valores logarítmicos uniformes muestreados aleatoriamente del espacio de valores logarítmicos permitidos. Si es mayor que 0, todos los límites deben ser finitos. Nótese que n_restarts_optimizer == 0 implica que se realiza una ejecución.
- normalize_ybool, default=False
Si los valores objetivo y están normalizados, la media y la varianza de los valores objetivo se establecen igual a 0 y 1 respectivamente. Esto se recomienda para los casos en los que se utilicen distribuciones a priori de media cero y varianza unitaria. Ten en cuenta que, en esta implementación, la normalización se invierte antes de que se informen las predicciones GP.
Distinto en la versión 0.23.
- copy_X_trainbool, default=True
Si es True, se almacena una copia persistente de los datos de entrenamiento en el objeto. De lo contrario, sólo se almacena una referencia a los datos de entrenamiento, lo que podría hacer que las predicciones cambien si los datos se modifican externamente.
- random_stateentero, instancia de RandomState o None, default=None
Determina la generación de números aleatorios utilizados para inicializar los centros. Se pasa un int para obtener resultados reproducibles a través de múltiples invocaciones a la función. Ver :term:
Glosario <random_state>.
- Atributos
- X_train_array-like de forma (n_samples, n_features) o lista de objetos
Vectores de características u otras representaciones de los datos de entrenamiento (también necesarios para la predicción).
- y_train_array-like de forma (n_samples,) o (n_samples, n_targets)
Valores objetivo en los datos de entrenamiento (también necesarios para la predicción)
- kernel_instancia de núcleo
El núcleo (kernel) utilizado para la predicción. La estructura del núcleo es la misma que la pasada como parámetro pero con hiperparámetros optimizados
- L_array-like de forma (n_samples, n_samples)
Descomposición de Cholesky triangular inferior del núcleo en
X_train_- alpha_array-like de forma (n_samples,)
Coeficientes duales de los puntos de datos de entrenamiento en el espacio del núcleo
- log_marginal_likelihood_value_float
La verosimilitud logarítmica marginal de
self.kernel_.theta
Ejemplos
>>> from sklearn.datasets import make_friedman2 >>> from sklearn.gaussian_process import GaussianProcessRegressor >>> from sklearn.gaussian_process.kernels import DotProduct, WhiteKernel >>> X, y = make_friedman2(n_samples=500, noise=0, random_state=0) >>> kernel = DotProduct() + WhiteKernel() >>> gpr = GaussianProcessRegressor(kernel=kernel, ... random_state=0).fit(X, y) >>> gpr.score(X, y) 0.3680... >>> gpr.predict(X[:2,:], return_std=True) (array([653.0..., 592.1...]), array([316.6..., 316.6...]))
Métodos
Ajusta el modelo de regresión del proceso gaussiano.
Obtiene los parámetros para este estimador.
Devuelve la verosimilitud logarítmica marginal de theta para los datos de entrenamiento.
Predice mediante el modelo de regresión del proceso gaussiano
Extrae muestras del proceso gaussiano y evalúa en X.
Devuelve el coeficiente de determinación \(R^2\) de la predicción.
Establece los parámetros de este estimador.
- fit()¶
Ajusta el modelo de regresión del proceso gaussiano.
- Parámetros
- Xarray-like de forma (n_samples, n_features) o lista de objetos
Vectores de características u otras representaciones de los datos de entrenamiento.
- yarray-like de forma (n_samples,) o (n_samples, n_targets)
Valores objetivo
- Devuelve
- selfdevuelve una instancia de sí misma.
- get_params()¶
Obtiene los parámetros para este estimador.
- Parámetros
- deepbool, default=True
Si es True, devolverá los parámetros para este estimador y los subobjetos contenidos que son estimadores.
- Devuelve
- paramsdict
Nombres de parámetros mapeados a sus valores.
- log_marginal_likelihood()¶
Devuelve la verosimilitud logarítmica marginal de theta para los datos de entrenamiento.
- Parámetros
- thetaarray-like de forma (n_kernel_params,) default=None
Hiperparámetros del núcleo para los que se evalúa la verosimilitud logarítmica marginal. Si es None, se devuelve la log_marginal_likelihood precalculada de
self.kernel_.theta.- eval_gradientbool, default=False
Si es True, el gradiente de la verosimilitud logarítmica marginal con respecto a los hiperparámetros del núcleo en la posición theta se devuelve adicionalmente. Si es True, theta no debe ser None.
- clone_kernelbool, default=True
Si es True, se copia el atributo kernel. Si es False, el atributo kernel se modifica, pero puede dar lugar a una mejora del rendimiento.
- Devuelve
- log_likelihoodfloat
Verosimilitud logarítmica marginal de theta para los datos de entrenamiento.
- log_likelihood_gradientndarray de forma (n_kernel_params,), optional
Gradiente de la verosimilitud logarítmica marginal con respecto a los hiperparámetros del núcleo en la posición theta. Sólo se devuelve cuando eval_gradient es True.
- predict()¶
Predice mediante el modelo de regresión del proceso gaussiano
También podemos predecir basándonos en un modelo no ajustado utilizando a priori un GP. Además de la media de la distribución predictiva, también su desviación estándar (return_std=True) o covarianza (return_cov=True). Ten en cuenta que como máximo se puede solicitar una de las dos.
- Parámetros
- Xarray-like de forma (n_samples, n_features) o lista de objetos
Puntos de consulta donde se evalúa el GP.
- return_stdbool, default=False
Si es True, se devuelve la desviación estándar de la distribución predictiva en los puntos de consulta junto con la media.
- return_covbool, default=False
Si es True, se devuelve la covarianza de la distribución predictiva conjunta en los puntos de consulta junto con la media.
- Devuelve
- y_meanndarray de forma (n_samples, [n_output_dims])
Media de la distribución predictiva de los puntos de consulta.
- y_stdndarray de forma (n_samples,), opcional
Desviación estándar de la distribución predictiva en los puntos de consulta. Sólo se devuelve cuando
return_stdes True.- y_covndarray de forma (n_samples, n_samples), optional
Covarianza de la distribución predictiva conjunta de los puntos de la consulta. Sólo se devuelve cuando
return_coves True.
- sample_y()¶
Extrae muestras del proceso gaussiano y evalúa en X.
- Parámetros
- Xarray-like de forma (n_samples, n_features) o lista de objetos
Puntos de consulta donde se evalúa el GP.
- n_samplesint, default=1
El número de muestras extraídas del proceso gaussiano
- random_stateentero, instancia de RandomState o None, default=0
Determina la generación de números aleatorios para extraer muestras al azar. Pasa un int para obtener resultados reproducibles a través de múltiples llamadas a la función. Ver :term:
Glosario <random_state>.
- Devuelve
- y_samplesndarray de forma (n_samples_X, [n_output_dims], n_samples)
Valores de n_muestras extraídos del proceso gaussiano y evaluados en los puntos de consulta.
- score()¶
Devuelve el coeficiente de determinación \(R^2\) de la predicción.
El coeficiente \(R^2\) se define como \((1 - \frac{u}{v})\), donde \(u\) es la suma residual de cuadrados
((y_true - y_pred) ** 2).sum()y \(v\) es la suma total de cuadrados((y_true - y_true.mean()) ** 2).sum(). La mejor puntuación posible es 1.0 y puede ser negativa (porque el modelo puede ser arbitrariamente peor). Un modelo constante que siempre predice el valor esperado dey, sin tener en cuenta las características de entrada, obtendría una puntuación \(R^2\) de 0,0.- Parámetros
- Xarray-like de forma (n_samples, n_features)
Muestras de prueba. Para algunos estimadores puede ser una matriz de núcleo precalculada o una lista de objetos genéricos con forma
(n_samples, n_samples_fitted), donden_samples_fittedes el número de muestras utilizadas en el ajuste para el estimador.- yarray-like de forma (n_samples,) o (n_samples, n_outputs)
Valores verdaderos para
X.- sample_weightarray-like de forma (n_samples,), default=None
Ponderaciones de la muestra.
- Devuelve
- scorefloat
\(R^2\) de
self.predict(X)con respecto ay.
Notas
La puntuación \(R^2\) utilizada al llamar a
scoreen un regresor utilizamultioutput='uniform_average'desde la versión 0.23 para mantener la coherencia con el valor predeterminado der2_score`. Esto influye en el métodoscorede todos los regresores de salida múltiple (excepto paraMultiOutputRegressor).
- set_params()¶
Establece los parámetros de este estimador.
El método funciona tanto en estimadores simples como en objetos anidados (como
Pipeline). Estos últimos tienen parámetros de la forma<component>__<parameter>`para que sea posible actualizar cada componente de un objeto anidado.- Parámetros
- **paramsdict
Parámetros del estimador.
- Devuelve
- selfinstancia del estimador
Instancia del estimador.