Nota
Haz clic aquí para descargar el código de ejemplo completo o para ejecutar este ejemplo en tu navegador a través de Binder
Aspectos Destacados de scikit-learn 0.22¶
¡Nos complace anunciar el lanzamiento de scikit-learn 0.22, que viene con muchas correcciones de errores y nuevas características¡. A continuación detallamos algunas de las principales características de esta versión. Para una lista exhaustiva de todos los cambios, por favor consulta la nota de lanzamiento.
Para instalar la última versión (con pip):
pip install --upgrade scikit-learn
o con conda:
conda install -c conda-forge scikit-learn
Nueva API plotting (de graficación)¶
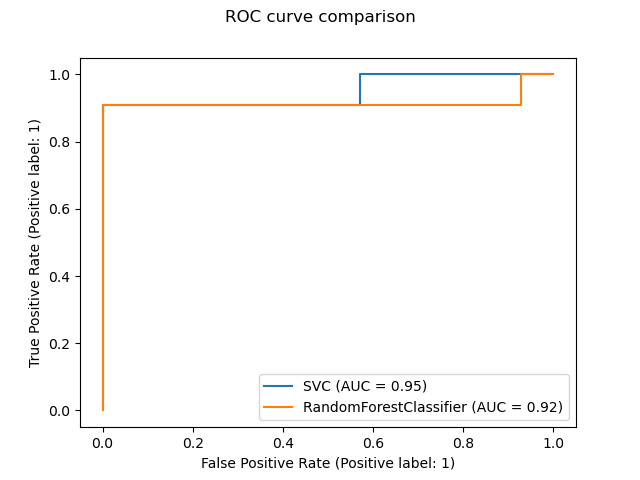
Una nueva API plotting está disponible para crear visualizaciones. Esta nueva API permite ajustar rápidamente el aspecto visual de un gráfico sin necesidad de volver a calcular. También es posible añadir diferentes trazados a la misma figura. El siguiente ejemplo ilustra plot_roc_curve, pero se admiten otras utilidades de graficación como plot_partial_dependence, plot_precision_recall_curve, y plot_confusion_matrix. Lee más sobre esta nueva API en el Manual de Usuario.
from sklearn.model_selection import train_test_split
from sklearn.svm import SVC
from sklearn.metrics import plot_roc_curve
from sklearn.ensemble import RandomForestClassifier
from sklearn.datasets import make_classification
import matplotlib.pyplot as plt
X, y = make_classification(random_state=0)
X_train, X_test, y_train, y_test = train_test_split(X, y, random_state=42)
svc = SVC(random_state=42)
svc.fit(X_train, y_train)
rfc = RandomForestClassifier(random_state=42)
rfc.fit(X_train, y_train)
svc_disp = plot_roc_curve(svc, X_test, y_test)
rfc_disp = plot_roc_curve(rfc, X_test, y_test, ax=svc_disp.ax_)
rfc_disp.figure_.suptitle("ROC curve comparison")
plt.show()
Clasificador y Regresor Apilados¶
StackingClassifier y StackingRegressor permiten tener una pila de estimadores con un clasificador final o un regresor. La generalización apilada consiste en apilar la salida de los estimadores individuales y utilizar un clasificador para calcular la predicción final. El apilamiento permite utilizar la fuerza de cada estimador individual utilizando su salida como entrada de un estimador final. Los estimadores base se ajustan a la totalidad de X, mientras que el estimador final se entrena utilizando las predicciones de validación cruzada de los estimadores base mediante cross_val_predict.
Lee más en el Manual de Usuario.
from sklearn.datasets import load_iris
from sklearn.svm import LinearSVC
from sklearn.linear_model import LogisticRegression
from sklearn.preprocessing import StandardScaler
from sklearn.pipeline import make_pipeline
from sklearn.ensemble import StackingClassifier
from sklearn.model_selection import train_test_split
X, y = load_iris(return_X_y=True)
estimators = [
('rf', RandomForestClassifier(n_estimators=10, random_state=42)),
('svr', make_pipeline(StandardScaler(),
LinearSVC(random_state=42)))
]
clf = StackingClassifier(
estimators=estimators, final_estimator=LogisticRegression()
)
X_train, X_test, y_train, y_test = train_test_split(
X, y, stratify=y, random_state=42
)
clf.fit(X_train, y_train).score(X_test, y_test)
Out:
0.9473684210526315
Importancia de las características basada en la permutación¶
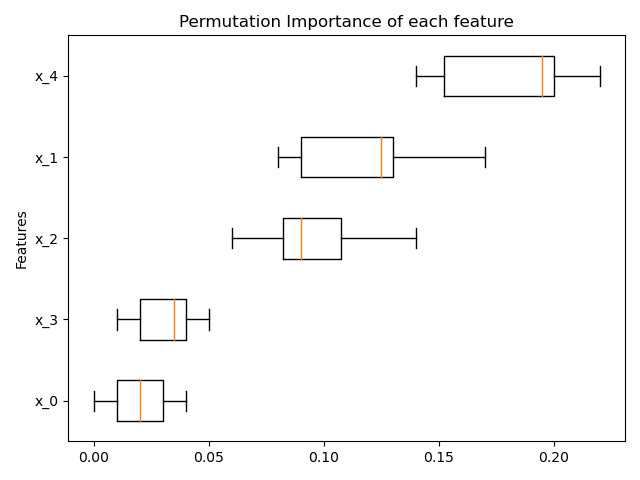
La inspection.permutation_importance puede utilizarse para obtener una estimación de la importancia de cada característica, para cualquier estimador ajustado:
import numpy as np
import matplotlib.pyplot as plt
from sklearn.datasets import make_classification
from sklearn.ensemble import RandomForestClassifier
from sklearn.inspection import permutation_importance
X, y = make_classification(random_state=0, n_features=5, n_informative=3)
feature_names = np.array([f'x_{i}' for i in range(X.shape[1])])
rf = RandomForestClassifier(random_state=0).fit(X, y)
result = permutation_importance(rf, X, y, n_repeats=10, random_state=0,
n_jobs=-1)
fig, ax = plt.subplots()
sorted_idx = result.importances_mean.argsort()
ax.boxplot(result.importances[sorted_idx].T,
vert=False, labels=feature_names[sorted_idx])
ax.set_title("Permutation Importance of each feature")
ax.set_ylabel("Features")
fig.tight_layout()
plt.show()
Soporte nativo de los valores faltantes para la potenciación del gradiente¶
El ensemble.HistGradientBoostingClassifier y ensemble.HistGradientBoostingRegressor tienen ahora soporte nativo para valores faltantes (NaN). Esto significa que no es necesario imputar los datos al entrenar o predecir.
from sklearn.experimental import enable_hist_gradient_boosting # noqa
from sklearn.ensemble import HistGradientBoostingClassifier
X = np.array([0, 1, 2, np.nan]).reshape(-1, 1)
y = [0, 0, 1, 1]
gbdt = HistGradientBoostingClassifier(min_samples_leaf=1).fit(X, y)
print(gbdt.predict(X))
Out:
[0 0 1 1]
Grafo disperso de vecinos más cercanos precalculados¶
La mayoría de los estimadores basados en gráficos de vecinos más cercanos ahora aceptan gráficos dispersos precalculados como entrada, para reutilizar el mismo gráfico para múltiples ajustes del estimador. Para utilizar esta función en un pipeline, se puede usar el parámetro memory, junto con uno de los dos nuevos transformadores, neighbors.KNeighborsTransformer y neighbors.RadiusNeighborsTransformer. El precalculo también puede ser realizado por estimadores personalizados para utilizar implementaciones alternativas, como los métodos de vecinos más cercanos aproximados. Consulta más detalles en el Manual de Usuario.
from tempfile import TemporaryDirectory
from sklearn.neighbors import KNeighborsTransformer
from sklearn.manifold import Isomap
from sklearn.pipeline import make_pipeline
X, y = make_classification(random_state=0)
with TemporaryDirectory(prefix="sklearn_cache_") as tmpdir:
estimator = make_pipeline(
KNeighborsTransformer(n_neighbors=10, mode='distance'),
Isomap(n_neighbors=10, metric='precomputed'),
memory=tmpdir)
estimator.fit(X)
# We can decrease the number of neighbors and the graph will not be
# recomputed.
estimator.set_params(isomap__n_neighbors=5)
estimator.fit(X)
Imputación Basada en KNN¶
Ahora se admite la imputación para completar los valores faltantes mediante k-Vecinos más Cercanos.
Los valores que faltan en cada muestra se imputan utilizando el valor medio de los n_neighbors vecinos más cercanos encontrados en el conjunto de entrenamiento. Dos muestras están cerca si las características que faltan están cerca. Por defecto, se utiliza una métrica de distancia euclidiana que admite valores faltantes, nan_euclidean_distances, se utiliza para encontrar los vecinos más cercanos.
Lee más en el Manual de Usuario.
from sklearn.impute import KNNImputer
X = [[1, 2, np.nan], [3, 4, 3], [np.nan, 6, 5], [8, 8, 7]]
imputer = KNNImputer(n_neighbors=2)
print(imputer.fit_transform(X))
Out:
[[1. 2. 4. ]
[3. 4. 3. ]
[5.5 6. 5. ]
[8. 8. 7. ]]
Poda de árboles¶
Ahora es posible podar la mayoría de los estimadores basados en árboles una vez construidos. La poda se basa en el coste-complejidad mínimo. Consulta más en el Manual de Usuario para más detalles.
X, y = make_classification(random_state=0)
rf = RandomForestClassifier(random_state=0, ccp_alpha=0).fit(X, y)
print("Average number of nodes without pruning {:.1f}".format(
np.mean([e.tree_.node_count for e in rf.estimators_])))
rf = RandomForestClassifier(random_state=0, ccp_alpha=0.05).fit(X, y)
print("Average number of nodes with pruning {:.1f}".format(
np.mean([e.tree_.node_count for e in rf.estimators_])))
Out:
Average number of nodes without pruning 22.3
Average number of nodes with pruning 6.4
Recuperar dataframes de OpenML¶
datasets.fetch_openml ahora puede devolver dataframe de pandas y así manejar adecuadamente conjuntos de datos con datos heterogéneos:
from sklearn.datasets import fetch_openml
titanic = fetch_openml('titanic', version=1, as_frame=True)
print(titanic.data.head()[['pclass', 'embarked']])
Out:
pclass embarked
0 1.0 S
1 1.0 S
2 1.0 S
3 1.0 S
4 1.0 S
Comprobación de la compatibilidad de un estimador con scikit-learn¶
Los desarrolladores pueden comprobar la compatibilidad de sus estimadores compatibles con scikit-learn utilizando check_estimator. Por ejemplo, el check_estimator(LinearSVC()) pasa.
Ahora proporcionamos un decorador específico de pytest que permite a pytest ejecutar todas las comprobaciones de forma independiente e informar de las comprobaciones que fallan.
- ..nota:
Esta entrada ha sido ligeramente actualizada en la versión 0.24, donde ya no se admite el paso de clases: en su lugar se pasan instancias.
from sklearn.linear_model import LogisticRegression
from sklearn.tree import DecisionTreeRegressor
from sklearn.utils.estimator_checks import parametrize_with_checks
@parametrize_with_checks([LogisticRegression(), DecisionTreeRegressor()])
def test_sklearn_compatible_estimator(estimator, check):
check(estimator)
La ROC AUC ahora admite la clasificación multiclase¶
La función roc_auc_score también puede utilizarse en la clasificación multiclase. Actualmente se admiten dos estrategias de promediación: el algoritmo uno-vs-uno calcula el promedio de las puntuaciones ROC AUC por pares, y el algoritmo uno-vs-resto calcula el promedio de las puntuaciones ROC AUC de cada clase frente a todas las demás clases. En ambos casos, las puntuaciones ROC AUC multiclase se calculan a partir de las estimaciones de probabilidad de que una muestra pertenezca a una clase concreta según el modelo. Los algoritmos OvO y OvR admiten la ponderación uniforme (average='macro') y la ponderación por la prevalencia (average='weighted').
Lee más en el Manual de Usuario.
from sklearn.datasets import make_classification
from sklearn.svm import SVC
from sklearn.metrics import roc_auc_score
X, y = make_classification(n_classes=4, n_informative=16)
clf = SVC(decision_function_shape='ovo', probability=True).fit(X, y)
print(roc_auc_score(y, clf.predict_proba(X), multi_class='ovo'))
Out:
0.9925707264957264
Tiempo total de ejecución del script: (0 minutos 1.892 segundos)
