sklearn.ensemble.IsolationForest¶
- class sklearn.ensemble.IsolationForest¶
Algoritmo de Bosque de Aislamiento.
Devuelve la puntuación de anomalía de cada muestra utilizando el algoritmo IsolationForest
El IsolationForest «aísla» las observaciones seleccionando aleatoriamente una característica y luego seleccionando aleatoriamente un valor de división entre los valores máximos y mínimos de la característica seleccionada.
Dado que la partición recursiva puede representarse mediante una estructura de árbol, el número de divisiones necesarias para aislar una muestra es equivalente a la longitud de la trayectoria desde el nodo raíz hasta el nodo final.
Esta longitud de la trayectoria, promediada sobre un bosque de tales árboles aleatorios, es una medida de normalidad y nuestra función de decisión.
La partición aleatoria produce trayectorias notablemente más cortas para las anomalías. Por lo tanto, cuando un bosque de árboles aleatorios produce colectivamente longitudes de trayectorias más cortas para muestras particulares, es muy probable que se trate de anomalías.
Leer más en el Manual de Usuario.
Nuevo en la versión 0.18.
- Parámetros
- n_estimatorsint, default=100
El número de estimadores base en el ensemble.
- max_samples«auto», int o float, default=»auto»
- El número de muestras a extraer de X para entrenar cada estimador base.
Si es int, entonces extrae
max_samplesmuestras.Si es float, entonces extrae
max_samples * X.shape[0]muestras.Si es «auto», entonces
max_samples=min(256, n_samples).
Si max_samples es mayor que el número de muestras proporcionado, se utilizarán todas las muestras para todos los árboles (sin muestreo).
- contamination“auto” o float, default=”auto”
La cantidad de contaminación del conjunto de datos, es decir, la proporción de valores atípicos en el conjunto de datos. Se utiliza al ajustar para definir el umbral en las puntuaciones de las muestras.
Si es «auto», el umbral se determina como en el documento original.
Si es float, la contaminación debe estar en el rango [0, 0.5].
Distinto en la versión 0.22: El valor predeterminado de
contaminationcambió de 0.1 a'auto'.- max_featuresint o float, default=1.0
El número de características a extraer de X para entrenar cada estimador base.
Si es int, entonces extrae
max_featurescaracterísticas.Si es float, entonces extrae
max_features * X.shape[1]características.
- bootstrapbool, default=False
Si es True, los árboles individuales se ajustan a subconjuntos aleatorios de los datos de entrenamiento muestreados con reemplazo. Si es False, se realiza un muestreo sin reemplazo.
- n_jobsint, default=None
El número de trabajos que se ejecutan en paralelo para
fitypredict.Nonesignifica 1 a menos que esté en un contextojoblib.parallel_backend.-1significa que se utilizan todos los procesadores. Ver Glosario para más detalles.- random_stateentero, instancia de RandomState o None, default=None
Controla la pseudo-aleatoriedad de la selección de los valores de las características y de división para cada paso de ramificación y cada árbol del bosque.
Pasa un número entero para que los resultados sean reproducibles a través de múltiples llamadas a la función. Ver Glosario.
- verboseint, default=0
Controla la verbosidad del proceso de construcción del árbol.
- warm_startbool, default=False
Cuando se establece como
True, reutiliza la solución de la llamada previa para ajustar y añadir más estimadores al ensemble, de lo contrario, sólo se ajusta un bosque nuevo. Ver Glosario.Nuevo en la versión 0.21.
- Atributos
- base_estimator_instancia ExtraTreeRegressor
La plantilla del estimador hijo utilizada para crear la colección de subestimadores ajustados.
- estimators_list de instancias ExtraTreeRegressor
La colección de subestimadores ajustados.
- estimators_features_list de ndarray
El subconjunto de características extraídas para cada estimador base.
estimators_samples_list de ndarrayEl subconjunto de muestras extraídas para cada estimador base.
- max_samples_int
El número real de muestras.
- offset_float
Desplazamiento utilizado para definir la función de decisión a partir de las puntuaciones brutas. Tenemos la relación:
decision_function = score_samples - offset_.offset_se define de la siguiente manera. Cuando el parámetro de contaminación se establece en «auto», el desplazamiento es igual a -0.5 ya que las puntuaciones de los valores típicos son cercanas a 0 y las puntuaciones de los valores atípicos son cercanas a -1. Cuando se proporciona un parámetro de contaminación diferente a «auto», el desplazamiento se define de tal manera que obtenemos el número esperado de valores atípicos (muestras con función de decisión < 0) en el entrenamiento.Nuevo en la versión 0.20.
- n_features_int
El número de características cuando se realiza el
fit.
Ver también
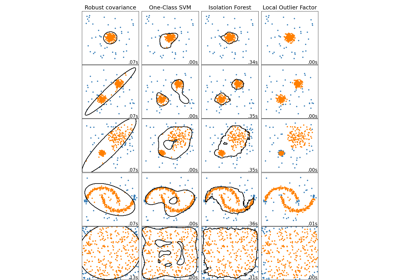
sklearn.covariance.EllipticEnvelopeUn objeto para detectar valores atípicos en un conjunto de datos con distribución Gaussiana.
sklearn.svm.OneClassSVMDetección No Supervisada de Valores Atípicos. Estima el soporte de una distribución de alta dimensión. La implementación se basa en libsvm.
sklearn.neighbors.LocalOutlierFactorDetección No Supervisada de Valores Atípicos mediante el Factor Local de Valores Atípicos (Local Outlier Factor, LOF).
Notas
La implementación se basa en un ensemble de ExtraTreeRegressor. La profundidad máxima de cada árbol se establece en
ceil(log_2(n))donde \(n\) es el número de muestras utilizadas para construir el árbol (ver (Liu et al., 2008) para más detalles).Referencias
- 1
Liu, Fei Tony, Ting, Kai Ming y Zhou, Zhi-Hua. «Isolation forest.» Data Mining, 2008. ICDM’08. Eighth IEEE International Conference on.
- 2
Liu, Fei Tony, Ting, Kai Ming y Zhou, Zhi-Hua. «Isolation-based anomaly detection.» ACM Transactions on Knowledge Discovery from Data (TKDD) 6.1 (2012): 3.
Ejemplos
>>> from sklearn.ensemble import IsolationForest >>> X = [[-1.1], [0.3], [0.5], [100]] >>> clf = IsolationForest(random_state=0).fit(X) >>> clf.predict([[0.1], [0], [90]]) array([ 1, 1, -1])
Métodos
Puntuación de anomalía media de X de los clasificadores base.
Estimador del ajuste.
Realiza el ajuste en X y devuelve las etiquetas para X.
Obtiene los parámetros para este estimador.
Predice si una muestra en particular es un valor atípico o no.
Lo contrario de la puntuación de anomalía definida en el documento original.
Establece los parámetros de este estimador.
- decision_function()¶
Puntuación de anomalía media de X de los clasificadores base.
La puntuación de anomalía de una muestra de entrada se calcula como la puntuación de anomalía media de los árboles del bosque.
La medida de normalidad de una observación dado un árbol es la profundidad de la hoja que contiene esta observación, que equivale al número de divisiones necesarias para aislar este punto. En caso de que haya varias observaciones n_left en la hoja, se añade la longitud media de la trayectoria de un árbol de aislamiento de muestras n_left.
- Parámetros
- X{array-like, sparse matrix} de forma (n_samples, n_features)
Las muestras de entrada. Internamente, se convertirá a
dtype=np.float32y si se proporciona una matriz dispersa, a unacsr_matrixdispersa.
- Devuelve
- scoresndarray de forma (n_samples,)
La puntuación de anomalía de las muestras de entrada. Cuanto más baja, más anormal. Las puntuaciones negativas representan valores atípicos, y las positivas, valores típicos.
- property estimators_samples_¶
El subconjunto de muestras extraídas para cada estimador base.
Devuelve una lista generada dinámicamente de índices que identifican las muestras utilizadas para el ajuste de cada miembro del ensemble, es decir, las muestras in-bag.
Nota: la lista se vuelve a crear en cada llamada a la propiedad para reducir la huella de memoria del objeto al no almacenar los datos de muestreo. Por lo tanto, la obtención de la propiedad puede ser más lenta de lo esperado.
- fit()¶
Estimador del ajuste.
- Parámetros
- X{array-like, sparse matrix} de forma (n_samples, n_features)
Las muestras de entrada. Utiliza
dtype=np.float32para obtener la máxima eficiencia. También se admiten matrices dispersas, utilizacsc_matrixdispersa para obtener la máxima eficiencia.- yIgnorado
No utilizado, está presente para la coherencia de la API por convención.
- sample_weightarray-like de forma (n_samples,), default=None
Ponderaciones de las muestras. Si es None, las muestras se ponderan por igual.
- Devuelve
- selfobject
Estimador ajustado.
- fit_predict()¶
Realiza el ajuste en X y devuelve las etiquetas para X.
Devuelve -1 para los valores atípicos y 1 para los valores típicos.
- Parámetros
- X{array-like, sparse matrix, dataframe} de forma (n_samples, n_features)
- yIgnorado
No utilizado, está presente para la coherencia de la API por convención.
- Devuelve
- yndarray de forma (n_samples,)
1 para los valores típicos, -1 para los valores atípicos.
- get_params()¶
Obtiene los parámetros para este estimador.
- Parámetros
- deepbool, default=True
Si es True, devolverá los parámetros para este estimador y los subobjetos contenidos que son estimadores.
- Devuelve
- paramsdict
Nombres de parámetros mapeados a sus valores.
- predict()¶
Predice si una muestra en particular es un valor atípico o no.
- Parámetros
- X{array-like, sparse matrix} de forma (n_samples, n_features)
Las muestras de entrada. Internamente, se convertirá a
dtype=np.float32y si se proporciona una matriz dispersa, a unacsr_matrixdispersa.
- Devuelve
- is_inlierndarray de forma (n_samples,)
Para cada observación, indica si debe considerarse o no (+1 o -1) como un valor típico según el modelo ajustado.
- score_samples()¶
Lo contrario de la puntuación de anomalía definida en el documento original.
La puntuación de anomalía de una muestra de entrada se calcula como la puntuación de anomalía media de los árboles del bosque.
La medida de normalidad de una observación dado un árbol es la profundidad de la hoja que contiene esta observación, que equivale al número de divisiones necesarias para aislar este punto. En caso de que haya varias observaciones n_left en la hoja, se añade la longitud media de la trayectoria de un árbol de aislamiento de muestras n_left.
- Parámetros
- X{array-like, sparse matrix} de forma (n_samples, n_features)
Las muestras de entrada.
- Devuelve
- scoresndarray de forma (n_samples,)
La puntuación de anomalía de las muestras de entrada. Cuanto más baja, más anormal.
- set_params()¶
Establece los parámetros de este estimador.
El método funciona tanto en estimadores simples como en objetos anidados (como
Pipeline). Estos últimos tienen parámetros de la forma<component>__<parameter>para que sea posible actualizar cada componente de un objeto anidado.- Parámetros
- **paramsdict
Parámetros del estimador.
- Devuelve
- selfinstancia del estimador
Instancia del estimador.