Nota
Haz clic aquí para descargar el código de ejemplo completo o para ejecutar este ejemplo en tu navegador a través de Binder
Aspectos Destacados de scikit-learn 0.23¶
¡Nos complace anunciar el lanzamiento de scikit-learn 0.23! Se han añadido muchas correcciones de errores y mejoras, así como algunas nuevas características clave. A continuación detallamos algunas de las principales características de esta versión. Para una lista exhaustiva de todos los cambios, por favor consulta las notas de publicación.
Para instalar la última versión (con pip):
pip install --upgrade scikit-learn
o con conda:
conda install -c conda-forge scikit-learn
Modelos Lineales Generalizados y pérdida de Poisson para la potenciación del gradiente¶
Ya están disponibles los tan esperados Modelos Lineales Generalizados con funciones de pérdida no normales. En particular, se han implementado tres nuevos regresores: PoissonRegressor, GammaRegressor, y TweedieRegressor. El regresor de Poisson puede utilizarse para modelar recuentos enteros positivos o frecuencias relativas. Lea más en el Manual de Usuario. Además, HistGradientBoostingRegressor también admite una nueva pérdida “poisson”.
import numpy as np
from sklearn.model_selection import train_test_split
from sklearn.linear_model import PoissonRegressor
from sklearn.experimental import enable_hist_gradient_boosting # noqa
from sklearn.ensemble import HistGradientBoostingRegressor
n_samples, n_features = 1000, 20
rng = np.random.RandomState(0)
X = rng.randn(n_samples, n_features)
# positive integer target correlated with X[:, 5] with many zeros:
y = rng.poisson(lam=np.exp(X[:, 5]) / 2)
X_train, X_test, y_train, y_test = train_test_split(X, y, random_state=rng)
glm = PoissonRegressor()
gbdt = HistGradientBoostingRegressor(loss='poisson', learning_rate=.01)
glm.fit(X_train, y_train)
gbdt.fit(X_train, y_train)
print(glm.score(X_test, y_test))
print(gbdt.score(X_test, y_test))
Out:
0.35776189065725783
0.42425183539869415
Representación visual de los estimadores¶
Los estimadores ahora pueden visualizarse en los cuadernos activando la opción display='diagram'. Esto es especialmente útil para resumir la estructura de los pipelines y otros estimadores compuestos, con interactividad para proporcionar detalles. Haz clic en la imagen de ejemplo que aparece a continuación para ampliar los elementos del pipeline. Consulta Visualización de estimadores compuestos para saber cómo puedes utilizar esta función.
from sklearn import set_config
from sklearn.pipeline import make_pipeline
from sklearn.preprocessing import OneHotEncoder, StandardScaler
from sklearn.impute import SimpleImputer
from sklearn.compose import make_column_transformer
from sklearn.linear_model import LogisticRegression
set_config(display='diagram')
num_proc = make_pipeline(SimpleImputer(strategy='median'), StandardScaler())
cat_proc = make_pipeline(
SimpleImputer(strategy='constant', fill_value='missing'),
OneHotEncoder(handle_unknown='ignore'))
preprocessor = make_column_transformer((num_proc, ('feat1', 'feat3')),
(cat_proc, ('feat0', 'feat2')))
clf = make_pipeline(preprocessor, LogisticRegression())
clf
Mejoras en la escalabilidad y estabilidad de KMeans¶
El estimador KMeans ha sido completamente reelaborado, y ahora es significativamente más rápido y estable. Además, el algoritmo de Elkan ahora es compatible con matrices dispersas. El estimador utiliza el paralelismo basado en OpenMP en lugar de depender de joblib, por lo que el parámetro n_jobs ya no tiene efecto. Para más detalles sobre cómo controlar el número de hilos (threads), consulta nuestras notas sobre Paralelismo.
import scipy
import numpy as np
from sklearn.model_selection import train_test_split
from sklearn.cluster import KMeans
from sklearn.datasets import make_blobs
from sklearn.metrics import completeness_score
rng = np.random.RandomState(0)
X, y = make_blobs(random_state=rng)
X = scipy.sparse.csr_matrix(X)
X_train, X_test, _, y_test = train_test_split(X, y, random_state=rng)
kmeans = KMeans(algorithm='elkan').fit(X_train)
print(completeness_score(kmeans.predict(X_test), y_test))
Out:
0.6585602198584783
Mejoras en los estimadores de Potenciación del Gradiente basados en histogramas¶
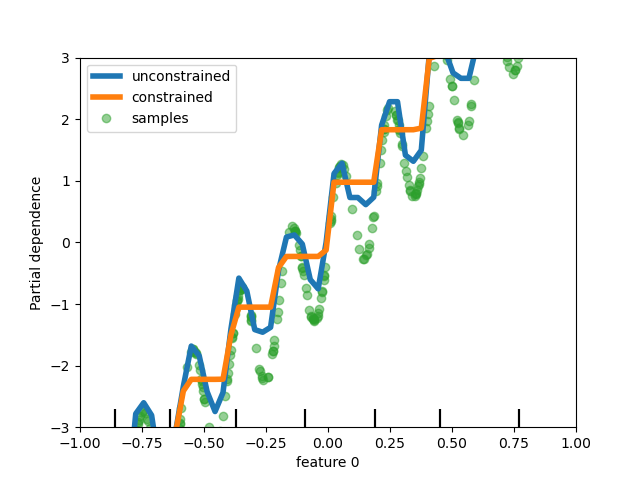
Se han realizado varias mejoras en HistGradientBoostingClassifier y HistGradientBoostingRegressor. Además de la pérdida de Poisson mencionada anteriormente, estos estimadores ahora admiten ponderaciones de muestra. También se ha añadido un criterio de parada temprana automática: la parada temprana se activa por defecto cuando el número de muestras supera los 10k. Por último, los usuarios ahora pueden definir restricciones monótonas para restringir las predicciones en función de las variaciones de características específicas. En el siguiente ejemplo, construimos un objetivo que generalmente está correlacionado positivamente con la primera característica, con algo de ruido. La aplicación de las restricciones monótonas permite que la predicción capture el efecto global de la primera característica, en lugar de ajustar el ruido.
import numpy as np
from matplotlib import pyplot as plt
from sklearn.model_selection import train_test_split
from sklearn.inspection import plot_partial_dependence
from sklearn.experimental import enable_hist_gradient_boosting # noqa
from sklearn.ensemble import HistGradientBoostingRegressor
n_samples = 500
rng = np.random.RandomState(0)
X = rng.randn(n_samples, 2)
noise = rng.normal(loc=0.0, scale=0.01, size=n_samples)
y = (5 * X[:, 0] + np.sin(10 * np.pi * X[:, 0]) - noise)
gbdt_no_cst = HistGradientBoostingRegressor().fit(X, y)
gbdt_cst = HistGradientBoostingRegressor(monotonic_cst=[1, 0]).fit(X, y)
disp = plot_partial_dependence(
gbdt_no_cst, X, features=[0], feature_names=['feature 0'],
line_kw={'linewidth': 4, 'label': 'unconstrained', "color": "tab:blue"})
plot_partial_dependence(gbdt_cst, X, features=[0],
line_kw={'linewidth': 4, 'label': 'constrained', "color": "tab:orange"},
ax=disp.axes_)
disp.axes_[0, 0].plot(
X[:, 0], y, 'o', alpha=.5, zorder=-1, label='samples', color="tab:green"
)
disp.axes_[0, 0].set_ylim(-3, 3); disp.axes_[0, 0].set_xlim(-1, 1)
plt.legend()
plt.show()
Admisión de ponderaciones de muestras para Lasso y ElasticNet¶
Los dos regresores lineales Lasso y ElasticNet ahora admiten ponderaciones de muestra.
from sklearn.model_selection import train_test_split
from sklearn.datasets import make_regression
from sklearn.linear_model import Lasso
import numpy as np
n_samples, n_features = 1000, 20
rng = np.random.RandomState(0)
X, y = make_regression(n_samples, n_features, random_state=rng)
sample_weight = rng.rand(n_samples)
X_train, X_test, y_train, y_test, sw_train, sw_test = train_test_split(
X, y, sample_weight, random_state=rng)
reg = Lasso()
reg.fit(X_train, y_train, sample_weight=sw_train)
print(reg.score(X_test, y_test, sw_test))
Out:
0.999791942438998
Tiempo total de ejecución del script: (0 minutos 1.663 segundos)
