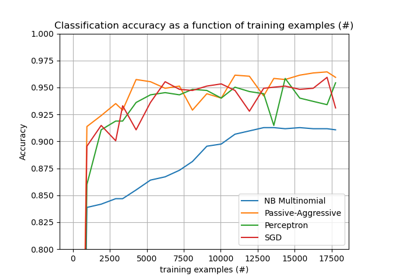
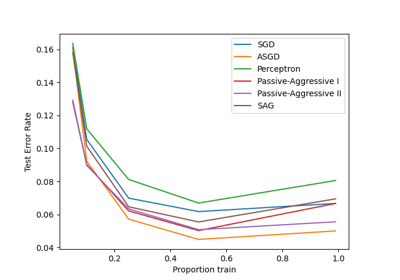
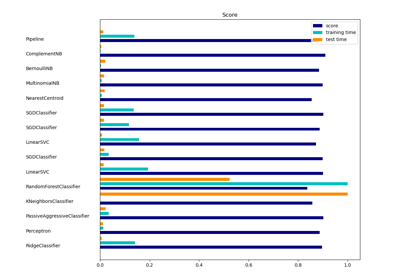
sklearn.linear_model.PassiveAggressiveClassifier¶
- class sklearn.linear_model.PassiveAggressiveClassifier¶
Clasificador agresivo pasivo (Passive Aggressive Classifier)
Más información en el Manual de usuario.
- Parámetros
- Cfloat, default=1.0
Tamaño máximo del paso o step (regularización). El valor predeterminado es 1.0.
- fit_interceptbool, default=True
Si el intercepto debe ser estimado o no. Si es False, se asume que los datos ya están centrados.
- max_iterint, default=1000
El número máximo de pasadas sobre los datos de entrenamiento (también conocido como épocas o epochs). Sólo afecta al comportamiento del método
fit, y no al métodopartial_fit.Nuevo en la versión 0.19.
- tolfloat o None, default=1e-3
El criterio de parada. Si no es None, las iteraciones se detendrán cuando (loss > previous_loss - tol).
Nuevo en la versión 0.19.
- early_stoppingbool, default=False
Indica si se utiliza la parada anticipada para finalizar el entrenamiento cuando la puntuación de validación no mejora. Si se establece en True, se reservará automáticamente una fracción estratificada de los datos de entrenamiento como validación y se finalizará el entrenamiento cuando la puntuación de validación no mejore en al menos tol para n_iter_no_change épocas consecutivas.
Nuevo en la versión 0.20.
- validation_fractionfloat, default=0.1
La proporción de los datos de entrenamiento que se reservan como conjunto de validación para la parada anticipada. Debe estar entre 0 y 1. Sólo se utiliza si early_stopping es True.
Nuevo en la versión 0.20.
- n_iter_no_changeint, default=5
Número de iteraciones sin mejora que hay que esperar antes de la parada anticipada.
Nuevo en la versión 0.20.
- shufflebool, default=True
Si los datos de entrenamiento deben ser aleatorizados o no después de cada época.
- verboseentero, default=0
Nivel de verbosidad
- losscadena de caracteres, default=»hinge»
La función de pérdida que se utilizará: hinge: equivalente a PA-I en el documento de referencia. squared_hinge: equivalente a PA-II en el documento de referencia.
- n_jobsint o None, default=None
El número de CPUs a utilizar para realizar el cálculo OVA (Uno Contra Todos, One Versus All, para problemas multiclase).
Nonesignifica 1 a menos que esté en un contextojoblib.parallel_backend.-1significa utilizar todos los procesadores. Ver Glosario para más detalles.- random_stateentero, instancia de RandomState, default=None
Se utiliza para reorganizar al azar los datos de entrenamiento, cuando
shufflese establece enTrue. Introduce un int para una salida reproducible a través de múltiples invocaciones de la función. Ver Glosario.- warm_startbool, default=False
Cuando se establece a
True, reutiliza la solución de la llamada anterior para ajustar como inicialización, de lo contrario, solamente borrará la solución anterior. Ver Glosario.Invocar repetidamente a fit o partial_fit cuando warm_start es True puede dar lugar a una solución diferente que cuando se invoca a fit una sola vez debido a la forma en que se mezclan al azar los datos.
- class_weightdict, {class_label: weight} or «balanced» or None, default=None
Preajuste para el parámetro de ajuste class_weight.
Ponderación asociada a las clases. Si no se da, se supone que todas las clases tienen ponderación uno.
El modo «balanced» utiliza los valores de y para ajustar automáticamente las ponderaciones inversamente proporcionales a las frecuencias de clase en los datos de entrada como
n_samples / (n_classes * np.bincount(y))Nuevo en la versión 0.17: parámetro class_weight para ponderar automáticamente las muestras.
- averagebool o int, default=False
Si se establece como True, calcula el promedio de los ponderados SGD en todas las actualizaciones y almacena el resultado en el atributo
coef_. Si se establece como un int mayor que 1, el promedio comenzará una vez que el número total de muestras vistas alcance elaverage. Así,average=10se empieza a promediar después de ver 10 muestras.Nuevo en la versión 0.19: parámetro average para utilizar el promedio de pesos en SGD
- Atributos
- coef_arreglo, forma = [1, n_features] si n_classes == 2 de lo contrario [n_classes, n_features]
Ponderaciones asignadas a las características.
- intercept_arreglo, forma = [1] si n_classes == 2 de lo contrario [n_classes]
Constantes en la función de decisión.
- n_iter_int
El número real de iteraciones para alcanzar el criterio de parada. Para los ajustes multiclase, es el máximo sobre cada ajuste binario.
- classes_arreglo de forma (n_classes,)
Etiquetas de clase únicas.
- t_int
Número de actualizaciones de peso realizadas durante el entrenamiento. Igual a
(n_iter_ * n_samples).- loss_function_invocable
Función de pérdida utilizada por el algoritmo.
Ver también
Referencias
Online Passive-Aggressive Algorithms <http://jmlr.csail.mit.edu/papers/volume7/crammer06a/crammer06a.pdf> K. Crammer, O. Dekel, J. Keshat, S. Shalev-Shwartz, Y. Singer - JMLR (2006)
Ejemplos
>>> from sklearn.linear_model import PassiveAggressiveClassifier >>> from sklearn.datasets import make_classification
>>> X, y = make_classification(n_features=4, random_state=0) >>> clf = PassiveAggressiveClassifier(max_iter=1000, random_state=0, ... tol=1e-3) >>> clf.fit(X, y) PassiveAggressiveClassifier(random_state=0) >>> print(clf.coef_) [[0.26642044 0.45070924 0.67251877 0.64185414]] >>> print(clf.intercept_) [1.84127814] >>> print(clf.predict([[0, 0, 0, 0]])) [1]
Métodos
Predecir las puntuaciones de confianza (confidence scores) de las muestras.
Convierte la matriz de coeficientes en formato de arreglo denso.
Ajuste del modelo lineal con el algoritmo pasivo-agresivo (Passive Aggresive algorithm).
Obtiene los parámetros para este estimador.
Ajuste del modelo lineal con el algoritmo pasivo-agresivo (Passive Aggresive algorithm).
Predice las etiquetas de clase para las muestras en X.
Devuelve la precisión media en los datos de prueba y las etiquetas dados.
Establece y validar los parámetros del estimador.
Convierte la matriz de coeficientes en formato disperso.
- decision_function()¶
Predecir las puntuaciones de confianza (confidence scores) de las muestras.
La puntuación de confianza de una muestra es proporcional a la distancia con signo de esa muestra al hiperplano.
- Parámetros
- Xarray-like o matriz dispersa, forma (n_samples, n_features)
Muestras.
- Devuelve
- arreglo, forma=(n_samples,) si n_classes == 2 de lo contrario (n_samples, n_classes)
Puntuaciones de confianza por combinación (muestra, clase). En el caso binario, la puntuación de confianza para self.classes_[1] donde >0 significa que esta clase sería predicha.
- densify()¶
Convierte la matriz de coeficientes en formato de arreglo denso.
Convierte el miembro
coef_(de vuelta) en un numpy.ndarray. Este es el formato predeterminado decoef_y se requiere para el ajuste, por lo que invocar este método sólo es necesario en los modelos que han sido previamente dispersados (sparsified); de lo contrario, es un no-op.- Devuelve
- self
Estimador ajustado.
- fit()¶
Ajuste del modelo lineal con el algoritmo pasivo-agresivo (Passive Aggresive algorithm).
- Parámetros
- X{array-like, sparse matrix} de forma (n_samples, n_features)
Datos del entrenamiento
- yarreglo de forma [n_samples]
Valores objetivo
- coef_initarreglo, forma = [n_classes,n_features]
Los coeficientes iniciales para iniciar la optimización en caliente (warm-start).
- intercept_initarreglo, forma = [n_classes]
La interceptación inicial para el arranque en caliente de la optimización.
- Devuelve
- selfdevuelve una instancia de sí misma.
- get_params()¶
Obtiene los parámetros para este estimador.
- Parámetros
- deepbool, default=True
Si es True, devolverá los parámetros para este estimador y los subobjetos contenidos que son estimadores.
- Devuelve
- paramsdict
Nombres de parámetros mapeados a sus valores.
- partial_fit()¶
Ajuste del modelo lineal con el algoritmo pasivo-agresivo (Passive Aggresive algorithm).
- Parámetros
- X{array-like, sparse matrix} de forma (n_samples, n_features)
Subconjunto de los datos de entrenamiento
- yarreglo de forma [n_samples]
Subconjunto de los valores objetivos
- classesarreglo, forma = [n_classes]
Clases a través de todas las invocaciones a partial_fit. Puede obtenerse mediante
np.unique(y_all), donde y_all es el vector objetivo de todo el conjunto de datos. Este argumento es necesario para la primera invocación a partial_fit y puede omitirse en las siguientes. Ten en cuenta que no es necesario que y contenga todas las etiquetas declasses.
- Devuelve
- selfdevuelve una instancia de sí misma.
- predict()¶
Predice las etiquetas de clase para las muestras en X.
- Parámetros
- Xarray-like o matriz dispersa, forma (n_samples, n_features)
Muestras.
- Devuelve
- Carreglo, forma [n_samples]
Etiqueta de clase predicha por muestra.
- score()¶
Devuelve la precisión media en los datos de prueba y las etiquetas dados.
En la clasificación multietiqueta, se trata de la precisión del subconjunto, que es una métrica rigurosa, ya que se requiere para cada muestra que cada conjunto de etiquetas sea predicho correctamente.
- Parámetros
- Xarray-like de forma (n_samples, n_features)
Muestras de prueba.
- yarray-like de forma (n_samples,) o (n_samples, n_outputs)
Etiquetas verdaderas para
X.- sample_weightarray-like de forma (n_samples,), default=None
Ponderaciones de muestra.
- Devuelve
- scorefloat
Precisión media de
self.predict(X)con relación ay.
- set_params()¶
Establece y validar los parámetros del estimador.
- Parámetros
- **kwargsdict
Parámetros del estimador.
- Devuelve
- selfobject
Instancia de estimador.
- sparsify()¶
Convierte la matriz de coeficientes en formato disperso.
Convierte el miembro
coef_en una matriz scipy.sparse, que para los modelos L1-regularizados puede ser mucho más eficiente en cuanto a memoria y almacenamiento que la representación numpy.ndarray habitual.El miembro
intercept_no se convierte.- Devuelve
- self
Estimador ajustado.
Notas
Para los modelos no dispersos, es decir, cuando no hay muchos ceros en
coef_, esto puede en realidad aumentar el uso de la memoria, así que utilice este método con cuidado. Una regla general es que el número de elementos cero, que puede ser calculado con(coef_ == 0).sum(), debe ser más del 50% para que esto proporcione beneficios significativos.Después de invocar a este método, el ajuste posterior con el método partial_fit (si lo hay) no funcionará hasta que llames a densify.