Nota
Haz clic aquí para descargar el código de ejemplo completo o para ejecutar este ejemplo en tu navegador a través de Binder
Selección de características basadas en modelos y secuenciales¶
Este ejemplo ilustra y compara dos enfoques para la selección de características: SelectFromModel que se basa en la importancia de las características, y SequentialFeatureSelection que se basa en un enfoque codicioso.
Utilizamos el conjunto de datos de Diabetes, que consiste en 10 características recolectadas de 442 pacientes de diabetes.
Autores: Manoj Kumar, Maria Telenczuk, Nicolas Hug.
Licencia: cláusula BSD 3
print(__doc__)
Cargando datos¶
Primero cargamos el conjunto de datos de diabetes que está disponible dentro de scikit-learn, e imprimimos su descripción:
from sklearn.datasets import load_diabetes
diabetes = load_diabetes()
X, y = diabetes.data, diabetes.target
print(diabetes.DESCR)
Out:
.. _diabetes_dataset:
Diabetes dataset
----------------
Ten baseline variables, age, sex, body mass index, average blood
pressure, and six blood serum measurements were obtained for each of n =
442 diabetes patients, as well as the response of interest, a
quantitative measure of disease progression one year after baseline.
**Data Set Characteristics:**
:Number of Instances: 442
:Number of Attributes: First 10 columns are numeric predictive values
:Target: Column 11 is a quantitative measure of disease progression one year after baseline
:Attribute Information:
- age age in years
- sex
- bmi body mass index
- bp average blood pressure
- s1 tc, T-Cells (a type of white blood cells)
- s2 ldl, low-density lipoproteins
- s3 hdl, high-density lipoproteins
- s4 tch, thyroid stimulating hormone
- s5 ltg, lamotrigine
- s6 glu, blood sugar level
Note: Each of these 10 feature variables have been mean centered and scaled by the standard deviation times `n_samples` (i.e. the sum of squares of each column totals 1).
Source URL:
https://www4.stat.ncsu.edu/~boos/var.select/diabetes.html
For more information see:
Bradley Efron, Trevor Hastie, Iain Johnstone and Robert Tibshirani (2004) "Least Angle Regression," Annals of Statistics (with discussion), 407-499.
(https://web.stanford.edu/~hastie/Papers/LARS/LeastAngle_2002.pdf)
Importancia de la función de los coeficientes¶
Para tener una idea de la importancia de las características, vamos a utilizar el estimador LassoCV. Las características con el mayor valor absoluto de coef_ se consideran las más importantes. Podemos observar los coeficientes directamente sin necesidad de escalarlos (o de escalar los datos) porque, por la descripción anterior, sabemos que las características ya estaban estandarizadas. Para un ejemplo más completo sobre las interpretaciones de los coeficientes de los modelos lineales, puede consultar Errores comunes en la interpretación de los coeficientes de los modelos lineales.
import matplotlib.pyplot as plt
import numpy as np
from sklearn.linear_model import LassoCV
lasso = LassoCV().fit(X, y)
importance = np.abs(lasso.coef_)
feature_names = np.array(diabetes.feature_names)
plt.bar(height=importance, x=feature_names)
plt.title("Feature importances via coefficients")
plt.show()
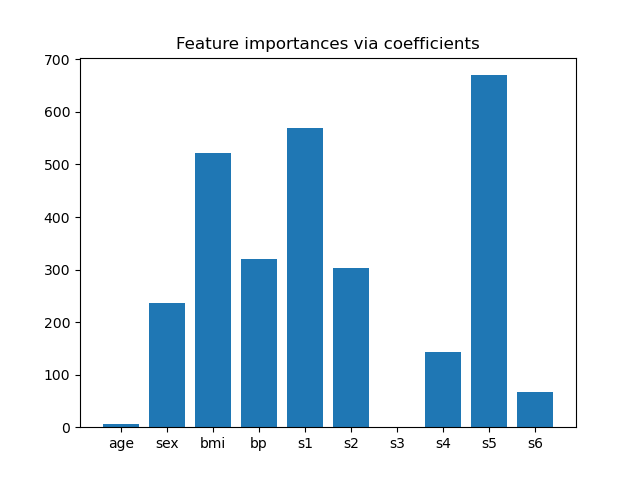
Seleccionar características basadas en la importancia¶
Ahora queremos seleccionar las dos características más importantes según los coeficientes. La SelectFromModel está pensada precisamente para eso. SelectFromModel acepta un parámetro threshold y seleccionará las características cuya importancia (definida por los coeficientes) esté por encima de este umbral.
Puesto que sólo queremos seleccionar 2 características, estableceremos este umbral ligeramente por encima del coeficiente de la tercera característica más importante.
from sklearn.feature_selection import SelectFromModel
from time import time
threshold = np.sort(importance)[-3] + 0.01
tic = time()
sfm = SelectFromModel(lasso, threshold=threshold).fit(X, y)
toc = time()
print("Features selected by SelectFromModel: "
f"{feature_names[sfm.get_support()]}")
print(f"Done in {toc - tic:.3f}s")
Out:
Features selected by SelectFromModel: ['s1' 's5']
Done in 0.066s
Seleccionar características con Selección de Características Secuenciales¶
Otra forma de seleccionar características es utilizar SequentialFeatureSelector (SFS). El SFS es un procedimiento codicioso en el que, en cada iteración, elegimos la mejor característica nueva para añadirla a nuestras características seleccionadas basándonos en una puntuación de validación cruzada. Es decir, empezamos con 0 características y elegimos la mejor característica individual con la mayor puntuación. El procedimiento se repite hasta alcanzar el número deseado de características seleccionadas.
También podemos ir en la dirección inversa (SFS hacia atrás), *p. ej. empieza con todas las características y escoge con codicia características para eliminar una por una. Ilustramos ambos enfoques aquí.
from sklearn.feature_selection import SequentialFeatureSelector
tic_fwd = time()
sfs_forward = SequentialFeatureSelector(lasso, n_features_to_select=2,
direction='forward').fit(X, y)
toc_fwd = time()
tic_bwd = time()
sfs_backward = SequentialFeatureSelector(lasso, n_features_to_select=2,
direction='backward').fit(X, y)
toc_bwd = time()
print("Features selected by forward sequential selection: "
f"{feature_names[sfs_forward.get_support()]}")
print(f"Done in {toc_fwd - tic_fwd:.3f}s")
print("Features selected by backward sequential selection: "
f"{feature_names[sfs_backward.get_support()]}")
print(f"Done in {toc_bwd - tic_bwd:.3f}s")
Out:
Features selected by forward sequential selection: ['bmi' 's5']
Done in 4.754s
Features selected by backward sequential selection: ['bmi' 's5']
Done in 14.278s
Discusión¶
Curiosamente, la selección hacia adelante y hacia atrás han seleccionado el mismo conjunto de características. En general, este no es el caso y los dos métodos llevarían a resultados diferentes.
También observamos que las características seleccionadas por SFS difieren de las seleccionadas por la importancia de las características: SFS selecciona bmi en lugar de s1. Sin embargo, esto suena razonable, ya que bmi corresponde a la tercera característica más importante de acuerdo con los coeficientes. Es bastante curioso si tenemos en cuenta que el SFS no hace uso de los coeficientes en absoluto.
Para terminar, debemos señalar que SelectFromModel es significativamente más rápido que SFS. De hecho, SelectFromModel sólo necesita ajustar un modelo una vez, mientras que SFS necesita validar de forma cruzada muchos modelos diferentes para cada una de las iteraciones. Sin embargo, SFS funciona con cualquier modelo, mientras que SelectFromModel requiere que el estimador subyacente exponga un atributo coef_ o un atributo feature_importances_. El SFS hacia adelante es más rápido que el SFS hacia atrás porque sólo necesita realizar n_features_to_select = 2 iteraciones, mientras que el SFS hacia atrás necesita realizar n_features - n_features = 8 iteraciones.
Tiempo total de ejecución del script: (0 minutos 19.257 segundos)
