Nota
Haz clic aquí para descargar el código de ejemplo completo o para ejecutar este ejemplo en tu navegador a través de Binder
Mapear datos a una distribución normal¶
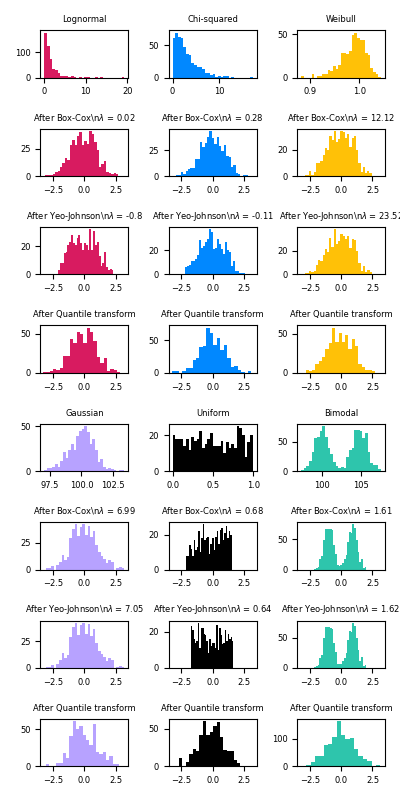
Este ejemplo demuestra el uso de las transformas Box-Cox y Yeo-Johnson a través de PowerTransformer para mapear los datos de varias distribuciones a una distribución normal.
La transformada de potencia es útil como transformación en problemas de modelado en los que se desea la homocedasticidad y la normalidad. A continuación se muestran ejemplos de Box-Cox y Yeo-Johnwon aplicados a seis distribuciones de probabilidad diferentes: Lognormal, Chi-cuadrado, Weibull, Gaussiana, Uniforme y Bimodal.
Ten en cuenta que las transformaciones asignan con éxito los datos a una distribución normal cuando se aplican a ciertos conjuntos de datos, pero son ineficaces con otros. Esto subraya la importancia de visualizar los datos antes y después de la transformación.
También ten en cuenta que aunque Box-Cox parece tener un mejor desempeño que Yeo-Johnson para las distribuciones lognormal y chi-cuadrado, ten en cuenta que Box-Cox no soporta entradas con valores negativos.
Para comparar, también sumamos la salida de QuantileTransformer. Puede forzar cualquier distribución arbitraria a un gaussiano, siempre que haya suficientes muestras de entrenamiento (millares). Debido a que es un método no paramétrico, es más difícil de interpretar que los parámetros (Box-Cox y Yeo-Johnson).
En conjuntos de datos «pequeños» (menos de unos cientos de puntos), la transformada de cuantil es propensa a sobreajustarse. Se recomienda entonces el uso de la transformada de potencia.
# Author: Eric Chang <ericchang2017@u.northwestern.edu>
# Nicolas Hug <contact@nicolas-hug.com>
# License: BSD 3 clause
import numpy as np
import matplotlib.pyplot as plt
from sklearn.preprocessing import PowerTransformer
from sklearn.preprocessing import QuantileTransformer
from sklearn.model_selection import train_test_split
print(__doc__)
N_SAMPLES = 1000
FONT_SIZE = 6
BINS = 30
rng = np.random.RandomState(304)
bc = PowerTransformer(method='box-cox')
yj = PowerTransformer(method='yeo-johnson')
# n_quantiles is set to the training set size rather than the default value
# to avoid a warning being raised by this example
qt = QuantileTransformer(n_quantiles=500, output_distribution='normal',
random_state=rng)
size = (N_SAMPLES, 1)
# lognormal distribution
X_lognormal = rng.lognormal(size=size)
# chi-squared distribution
df = 3
X_chisq = rng.chisquare(df=df, size=size)
# weibull distribution
a = 50
X_weibull = rng.weibull(a=a, size=size)
# gaussian distribution
loc = 100
X_gaussian = rng.normal(loc=loc, size=size)
# uniform distribution
X_uniform = rng.uniform(low=0, high=1, size=size)
# bimodal distribution
loc_a, loc_b = 100, 105
X_a, X_b = rng.normal(loc=loc_a, size=size), rng.normal(loc=loc_b, size=size)
X_bimodal = np.concatenate([X_a, X_b], axis=0)
# create plots
distributions = [
('Lognormal', X_lognormal),
('Chi-squared', X_chisq),
('Weibull', X_weibull),
('Gaussian', X_gaussian),
('Uniform', X_uniform),
('Bimodal', X_bimodal)
]
colors = ['#D81B60', '#0188FF', '#FFC107',
'#B7A2FF', '#000000', '#2EC5AC']
fig, axes = plt.subplots(nrows=8, ncols=3, figsize=plt.figaspect(2))
axes = axes.flatten()
axes_idxs = [(0, 3, 6, 9), (1, 4, 7, 10), (2, 5, 8, 11), (12, 15, 18, 21),
(13, 16, 19, 22), (14, 17, 20, 23)]
axes_list = [(axes[i], axes[j], axes[k], axes[l])
for (i, j, k, l) in axes_idxs]
for distribution, color, axes in zip(distributions, colors, axes_list):
name, X = distribution
X_train, X_test = train_test_split(X, test_size=.5)
# perform power transforms and quantile transform
X_trans_bc = bc.fit(X_train).transform(X_test)
lmbda_bc = round(bc.lambdas_[0], 2)
X_trans_yj = yj.fit(X_train).transform(X_test)
lmbda_yj = round(yj.lambdas_[0], 2)
X_trans_qt = qt.fit(X_train).transform(X_test)
ax_original, ax_bc, ax_yj, ax_qt = axes
ax_original.hist(X_train, color=color, bins=BINS)
ax_original.set_title(name, fontsize=FONT_SIZE)
ax_original.tick_params(axis='both', which='major', labelsize=FONT_SIZE)
for ax, X_trans, meth_name, lmbda in zip(
(ax_bc, ax_yj, ax_qt),
(X_trans_bc, X_trans_yj, X_trans_qt),
('Box-Cox', 'Yeo-Johnson', 'Quantile transform'),
(lmbda_bc, lmbda_yj, None)):
ax.hist(X_trans, color=color, bins=BINS)
title = 'After {}'.format(meth_name)
if lmbda is not None:
title += r'\n$\lambda$ = {}'.format(lmbda)
ax.set_title(title, fontsize=FONT_SIZE)
ax.tick_params(axis='both', which='major', labelsize=FONT_SIZE)
ax.set_xlim([-3.5, 3.5])
plt.tight_layout()
plt.show()
Tiempo total de ejecución del script: (0 minutos 1.810 segundos)
