Nota
Haz clic aquí para descargar el código completo del ejemplo o para ejecutar este ejemplo en tu navegador a través de Binder
Validación cruzada anidada y no anidada¶
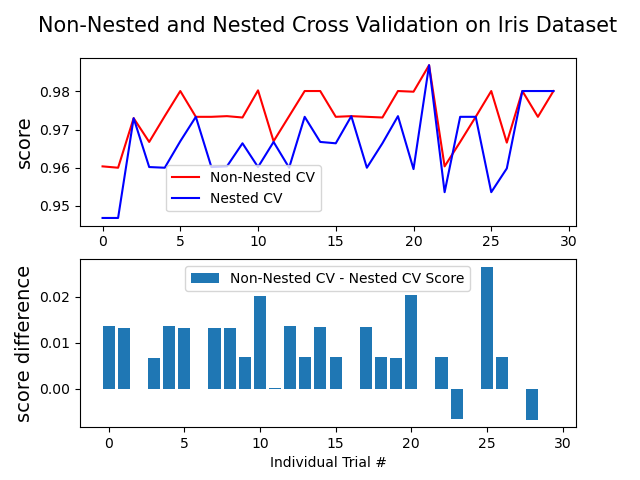
Este ejemplo compara las estrategias de validación cruzada anidada y no anidada en un clasificador del conjunto de datos iris. La validación cruzada anidada (CV) se utiliza a menudo para entrenar un modelo en el que también hay que optimizar los hiperparámetros. La CV anidada estima el error de generalización del modelo subyacente y su búsqueda de (hiper)parámetros. La elección de los parámetros que maximizan la CV no anidada sesga el modelo hacia el conjunto de datos, lo que produce una puntuación demasiado optimista.
La selección de modelos sin CV anidada utiliza los mismos datos para ajustar los parámetros del modelo y evaluar su rendimiento. Así, la información puede «filtrarse» en el modelo y sobreajustar los datos. La magnitud de este efecto depende principalmente del tamaño del conjunto de datos y de la estabilidad del modelo. Ver Cawley y Talbot 1 para un análisis de estas incidencias.
Para evitar este problema, la CV anidada utiliza efectivamente una serie de divisiones de conjuntos de entrenamiento/validación/prueba. En el bucle interno (aquí ejecutado por GridSearchCV), la puntuación se maximiza aproximadamente ajustando un modelo a cada conjunto de entrenamiento, y luego se maximiza directamente en la selección de (hiper)parámetros sobre el conjunto de validación. En el bucle externo (aquí en cross_val_score), el error de generalización se estima promediando las puntuaciones de los conjuntos de prueba en varias divisiones de conjuntos de datos.
El siguiente ejemplo utiliza un clasificador de vectores de soporte con un kernel no lineal para construir un modelo con hiperparámetros optimizados mediante la búsqueda en cuadrícula. Comparamos el rendimiento de las estrategias de CV no anidada y anidada tomando la diferencia entre sus puntuaciones.
See Also:
Referencias:
Out:
Average difference of 0.007581 with std. dev. of 0.007833.
from sklearn.datasets import load_iris
from matplotlib import pyplot as plt
from sklearn.svm import SVC
from sklearn.model_selection import GridSearchCV, cross_val_score, KFold
import numpy as np
print(__doc__)
# Number of random trials
NUM_TRIALS = 30
# Load the dataset
iris = load_iris()
X_iris = iris.data
y_iris = iris.target
# Set up possible values of parameters to optimize over
p_grid = {"C": [1, 10, 100],
"gamma": [.01, .1]}
# We will use a Support Vector Classifier with "rbf" kernel
svm = SVC(kernel="rbf")
# Arrays to store scores
non_nested_scores = np.zeros(NUM_TRIALS)
nested_scores = np.zeros(NUM_TRIALS)
# Loop for each trial
for i in range(NUM_TRIALS):
# Choose cross-validation techniques for the inner and outer loops,
# independently of the dataset.
# E.g "GroupKFold", "LeaveOneOut", "LeaveOneGroupOut", etc.
inner_cv = KFold(n_splits=4, shuffle=True, random_state=i)
outer_cv = KFold(n_splits=4, shuffle=True, random_state=i)
# Non_nested parameter search and scoring
clf = GridSearchCV(estimator=svm, param_grid=p_grid, cv=inner_cv)
clf.fit(X_iris, y_iris)
non_nested_scores[i] = clf.best_score_
# Nested CV with parameter optimization
nested_score = cross_val_score(clf, X=X_iris, y=y_iris, cv=outer_cv)
nested_scores[i] = nested_score.mean()
score_difference = non_nested_scores - nested_scores
print("Average difference of {:6f} with std. dev. of {:6f}."
.format(score_difference.mean(), score_difference.std()))
# Plot scores on each trial for nested and non-nested CV
plt.figure()
plt.subplot(211)
non_nested_scores_line, = plt.plot(non_nested_scores, color='r')
nested_line, = plt.plot(nested_scores, color='b')
plt.ylabel("score", fontsize="14")
plt.legend([non_nested_scores_line, nested_line],
["Non-Nested CV", "Nested CV"],
bbox_to_anchor=(0, .4, .5, 0))
plt.title("Non-Nested and Nested Cross Validation on Iris Dataset",
x=.5, y=1.1, fontsize="15")
# Plot bar chart of the difference.
plt.subplot(212)
difference_plot = plt.bar(range(NUM_TRIALS), score_difference)
plt.xlabel("Individual Trial #")
plt.legend([difference_plot],
["Non-Nested CV - Nested CV Score"],
bbox_to_anchor=(0, 1, .8, 0))
plt.ylabel("score difference", fontsize="14")
plt.show()
Tiempo total de ejecución del script: (0 minutos 9.320 segundos)
