Nota
Haz clic aquí para descargar el código de ejemplo completo o para ejecutar este ejemplo en tu navegador a través de Binder
Selección de características univariantes¶
Un ejemplo que muestra la selección univariante de características.
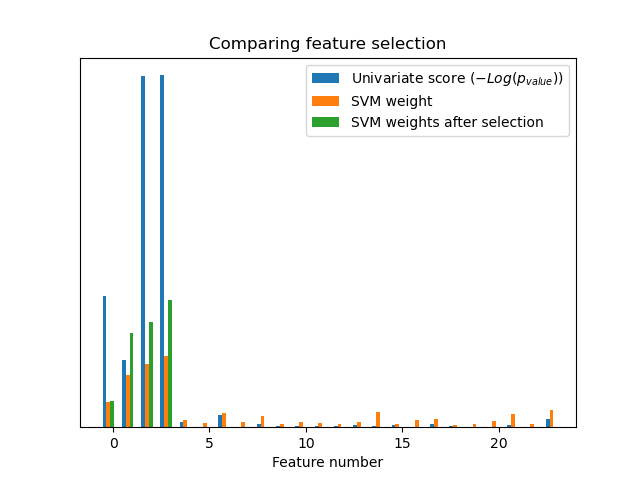
Se añaden características ruidosas (no informativas) a los datos del iris y se aplica la selección de características univariantes. Para cada característica, trazamos los valores p para la selección de características univariantes y los pesos correspondientes de una SVM. Podemos ver que la selección de rasgos univariantes selecciona los rasgos informativos y que éstos tienen mayores pesos de la SVM.
En el conjunto total de características, sólo las 4 primeras son significativas. Podemos ver que tienen la puntuación más alta con la selección de características univariantes. La SVM asigna un gran peso a una de estas características, pero también selecciona muchas de las características no informativas. La aplicación de la selección de características univariantes antes de la SVM aumenta el peso de la SVM atribuido a las características significativas y, por tanto, mejorará la clasificación.
Out:
Classification accuracy without selecting features: 0.789
Classification accuracy after univariate feature selection: 0.868
print(__doc__)
import numpy as np
import matplotlib.pyplot as plt
from sklearn.datasets import load_iris
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import MinMaxScaler
from sklearn.svm import LinearSVC
from sklearn.pipeline import make_pipeline
from sklearn.feature_selection import SelectKBest, f_classif
# #############################################################################
# Import some data to play with
# The iris dataset
X, y = load_iris(return_X_y=True)
# Some noisy data not correlated
E = np.random.RandomState(42).uniform(0, 0.1, size=(X.shape[0], 20))
# Add the noisy data to the informative features
X = np.hstack((X, E))
# Split dataset to select feature and evaluate the classifier
X_train, X_test, y_train, y_test = train_test_split(
X, y, stratify=y, random_state=0
)
plt.figure(1)
plt.clf()
X_indices = np.arange(X.shape[-1])
# #############################################################################
# Univariate feature selection with F-test for feature scoring
# We use the default selection function to select the four
# most significant features
selector = SelectKBest(f_classif, k=4)
selector.fit(X_train, y_train)
scores = -np.log10(selector.pvalues_)
scores /= scores.max()
plt.bar(X_indices - .45, scores, width=.2,
label=r'Univariate score ($-Log(p_{value})$)')
# #############################################################################
# Compare to the weights of an SVM
clf = make_pipeline(MinMaxScaler(), LinearSVC())
clf.fit(X_train, y_train)
print('Classification accuracy without selecting features: {:.3f}'
.format(clf.score(X_test, y_test)))
svm_weights = np.abs(clf[-1].coef_).sum(axis=0)
svm_weights /= svm_weights.sum()
plt.bar(X_indices - .25, svm_weights, width=.2, label='SVM weight')
clf_selected = make_pipeline(
SelectKBest(f_classif, k=4), MinMaxScaler(), LinearSVC()
)
clf_selected.fit(X_train, y_train)
print('Classification accuracy after univariate feature selection: {:.3f}'
.format(clf_selected.score(X_test, y_test)))
svm_weights_selected = np.abs(clf_selected[-1].coef_).sum(axis=0)
svm_weights_selected /= svm_weights_selected.sum()
plt.bar(X_indices[selector.get_support()] - .05, svm_weights_selected,
width=.2, label='SVM weights after selection')
plt.title("Comparing feature selection")
plt.xlabel('Feature number')
plt.yticks(())
plt.axis('tight')
plt.legend(loc='upper right')
plt.show()
Tiempo total de ejecución del script: (0 minutos 0.166 segundos)
