Nota
Haz clic en aquí para descargar el código de ejemplo completo o para ejecutar este ejemplo en tu navegador a través de Binder
Comparación de la calibración de los clasificadores¶
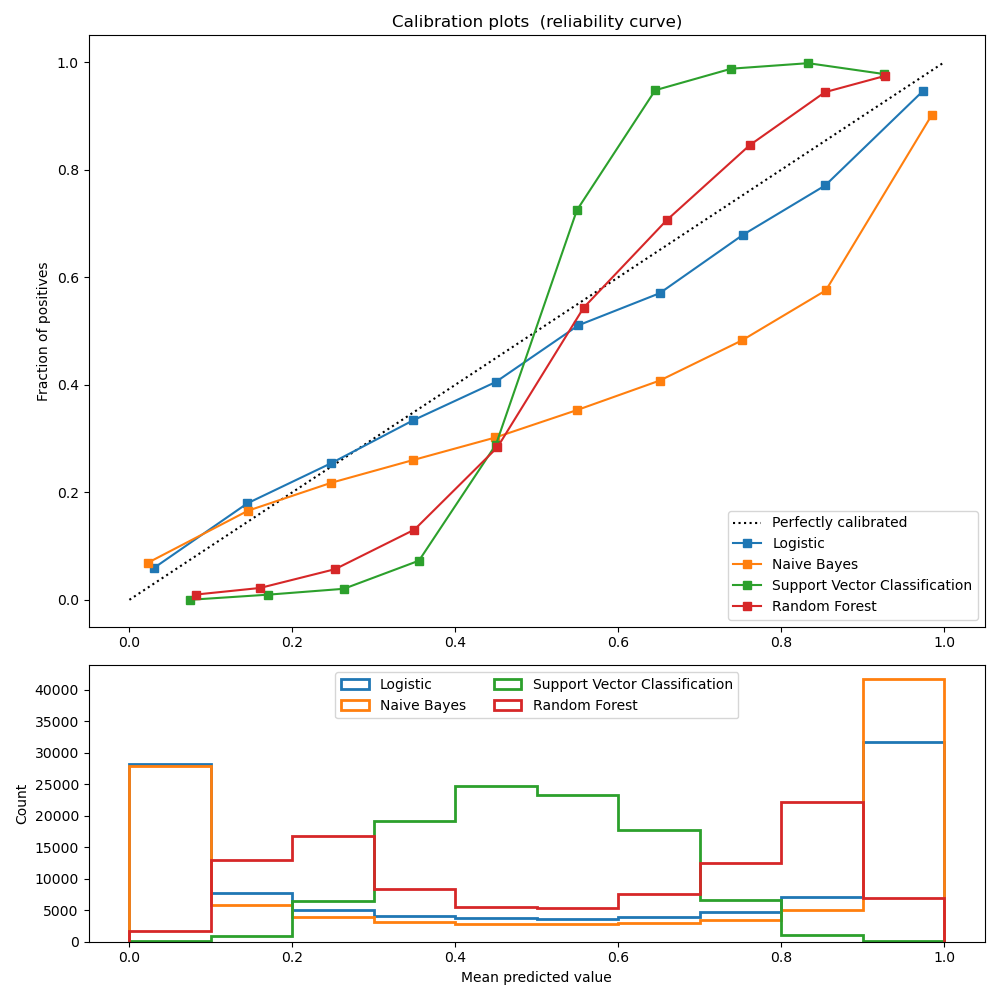
Los clasificadores bien calibrados son clasificadores probabilísticos para los que el resultado del método predict_proba puede interpretarse directamente como un nivel de confianza. Por ejemplo, un clasificador (binario) bien calibrado debería clasificar las muestras de tal manera que, entre las muestras a las que dio un valor de predict_proba cercano a 0,8, aproximadamente el 80% pertenecen realmente a la clase positiva.
LogisticRegression devuelve predicciones bien calibradas, ya que optimiza directamente la pérdida logarítmica. En cambio, los otros métodos devuelven probabilidades sesgadas, con diferentes sesgos por método:
GaussianNaiveBayes tiende a empujar las probabilidades a 0 o 1 (observe los recuentos en los histogramas). Esto se debe principalmente a que asume que las características son condicionalmente independientes dada la clase, lo que no es el caso en este conjunto de datos que contiene 2 características redundantes.
RandomForestClassifier muestra el comportamiento opuesto: los histogramas muestran picos con una probabilidad de aproximadamente 0,2 y 0,9, mientras que las probabilidades cercanas a 0 o 1 son muy raras. Niculescu-Mizil y Caruana 1 dan una explicación para esto «Los métodos como el bagging y los bosques aleatorios que promedian las predicciones de un conjunto de modelos base pueden tener dificultades para hacer predicciones cercanas a 0 y 1 porque la varianza en los modelos base subyacentes sesgará las predicciones que deberían estar cerca de cero o uno lejos de estos valores. Dado que las predicciones están restringidas al intervalo [0,1], los errores causados por la varianza tienden a ser unilaterales cerca de cero y uno. Por ejemplo, si un modelo debe predecir p = 0 para un caso, la única forma de conseguirlo es que todos los árboles empaquetados predigan cero. Si añadimos ruido a los árboles sobre los que el ensamblaje hace la media, este ruido hará que algunos árboles predigan valores mayores que 0 para este caso, alejando así la predicción media del conjunto ensamblado de 0. Observamos este efecto con más fuerza con los bosques aleatorios porque los árboles de nivel base entrenados con bosques aleatorios tienen una varianza relativamente alta debido al subconjunto de características». Como resultado, la curva de calibración muestra una forma sigmoidea característica, lo que indica que el clasificador podría confiar más en su «intuición» y devolver probabilidades más cercanas a 0 o 1 normalmente.
La clasificación de vectores de soporte (SVC) muestra una curva aún más sigmoidea que el clasificador RandomForest, lo que es típico de los métodos de margen máximo (compárese con Niculescu-Mizil y Caruana 1), que se centran en las muestras duras que están cerca de la frontera de decisión (los vectores de soporte).
Referencias:
print(__doc__)
# Author: Jan Hendrik Metzen <jhm@informatik.uni-bremen.de>
# License: BSD Style.
import numpy as np
np.random.seed(0)
import matplotlib.pyplot as plt
from sklearn import datasets
from sklearn.naive_bayes import GaussianNB
from sklearn.linear_model import LogisticRegression
from sklearn.ensemble import RandomForestClassifier
from sklearn.svm import LinearSVC
from sklearn.calibration import calibration_curve
X, y = datasets.make_classification(n_samples=100000, n_features=20,
n_informative=2, n_redundant=2)
train_samples = 100 # Samples used for training the models
X_train = X[:train_samples]
X_test = X[train_samples:]
y_train = y[:train_samples]
y_test = y[train_samples:]
# Create classifiers
lr = LogisticRegression()
gnb = GaussianNB()
svc = LinearSVC(C=1.0)
rfc = RandomForestClassifier()
# #############################################################################
# Plot calibration plots
plt.figure(figsize=(10, 10))
ax1 = plt.subplot2grid((3, 1), (0, 0), rowspan=2)
ax2 = plt.subplot2grid((3, 1), (2, 0))
ax1.plot([0, 1], [0, 1], "k:", label="Perfectly calibrated")
for clf, name in [(lr, 'Logistic'),
(gnb, 'Naive Bayes'),
(svc, 'Support Vector Classification'),
(rfc, 'Random Forest')]:
clf.fit(X_train, y_train)
if hasattr(clf, "predict_proba"):
prob_pos = clf.predict_proba(X_test)[:, 1]
else: # use decision function
prob_pos = clf.decision_function(X_test)
prob_pos = \
(prob_pos - prob_pos.min()) / (prob_pos.max() - prob_pos.min())
fraction_of_positives, mean_predicted_value = \
calibration_curve(y_test, prob_pos, n_bins=10)
ax1.plot(mean_predicted_value, fraction_of_positives, "s-",
label="%s" % (name, ))
ax2.hist(prob_pos, range=(0, 1), bins=10, label=name,
histtype="step", lw=2)
ax1.set_ylabel("Fraction of positives")
ax1.set_ylim([-0.05, 1.05])
ax1.legend(loc="lower right")
ax1.set_title('Calibration plots (reliability curve)')
ax2.set_xlabel("Mean predicted value")
ax2.set_ylabel("Count")
ax2.legend(loc="upper center", ncol=2)
plt.tight_layout()
plt.show()
Tiempo total de ejecución del script: (0 minutos 1.549 segundos)
