Nota
Haz clic aquí para descargar el código de ejemplo completo o para ejecutar este ejemplo en tu navegador a través de Binder
Traza diferentes clasificadores SVM en el conjunto de datos iris¶
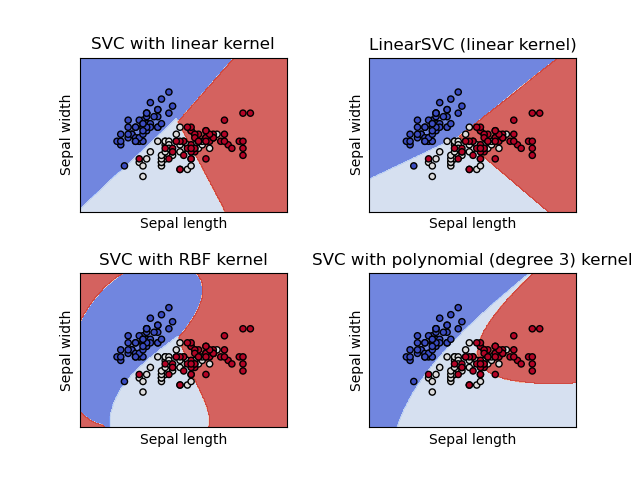
Comparación de diferentes clasificadores SVM lineales en una proyección 2D del conjunto de datos iris. Sólo tenemos en cuenta las dos primeras características de este conjunto de datos:
Longitud del sepal
Ancho del sepal
Este ejemplo muestra cómo trazar la superficie de decisión para cuatro clasificadores SVM con diferentes núcleos.
Los modelos lineales LinearSVC() y SVC(kernel='linear') producen límites de decisión ligeramente diferentes. Esto puede ser una consecuencia de las siguientes diferencias:
LinearSVCminimiza la pérdida de bisagra al cuadrado mientras queSVCminimiza la pérdida de bisagra regular.LinearSVCutiliza la reducción multiclase Uno-vs-Todos (también conocida como Uno-vs-Resto) mientras queSVCutiliza la reducción multiclase Uno-vs-Uno.
Ambos modelos lineales tienen límites de decisión lineales (hiperplano intersectorial) mientras que los modelos no lineales del núcleo (polinomio o Gaussian RBF) tienen límites de decisión no lineales más flexibles con formas que dependen del tipo de núcleo y sus parámetros.
Nota
si bien el trazado de la función de decisión de los clasificadores para conjuntos de datos 2D de juguete puede ayudar a obtener una comprensión intuitiva de su respectivo poder expresivo, ten en cuenta que esas intuiciones no siempre se generalizan a problemas más realistas de alta dimensión.
print(__doc__)
import numpy as np
import matplotlib.pyplot as plt
from sklearn import svm, datasets
def make_meshgrid(x, y, h=.02):
"""Create a mesh of points to plot in
Parameters
----------
x: data to base x-axis meshgrid on
y: data to base y-axis meshgrid on
h: stepsize for meshgrid, optional
Returns
-------
xx, yy : ndarray
"""
x_min, x_max = x.min() - 1, x.max() + 1
y_min, y_max = y.min() - 1, y.max() + 1
xx, yy = np.meshgrid(np.arange(x_min, x_max, h),
np.arange(y_min, y_max, h))
return xx, yy
def plot_contours(ax, clf, xx, yy, **params):
"""Plot the decision boundaries for a classifier.
Parameters
----------
ax: matplotlib axes object
clf: a classifier
xx: meshgrid ndarray
yy: meshgrid ndarray
params: dictionary of params to pass to contourf, optional
"""
Z = clf.predict(np.c_[xx.ravel(), yy.ravel()])
Z = Z.reshape(xx.shape)
out = ax.contourf(xx, yy, Z, **params)
return out
# import some data to play with
iris = datasets.load_iris()
# Take the first two features. We could avoid this by using a two-dim dataset
X = iris.data[:, :2]
y = iris.target
# we create an instance of SVM and fit out data. We do not scale our
# data since we want to plot the support vectors
C = 1.0 # SVM regularization parameter
models = (svm.SVC(kernel='linear', C=C),
svm.LinearSVC(C=C, max_iter=10000),
svm.SVC(kernel='rbf', gamma=0.7, C=C),
svm.SVC(kernel='poly', degree=3, gamma='auto', C=C))
models = (clf.fit(X, y) for clf in models)
# title for the plots
titles = ('SVC with linear kernel',
'LinearSVC (linear kernel)',
'SVC with RBF kernel',
'SVC with polynomial (degree 3) kernel')
# Set-up 2x2 grid for plotting.
fig, sub = plt.subplots(2, 2)
plt.subplots_adjust(wspace=0.4, hspace=0.4)
X0, X1 = X[:, 0], X[:, 1]
xx, yy = make_meshgrid(X0, X1)
for clf, title, ax in zip(models, titles, sub.flatten()):
plot_contours(ax, clf, xx, yy,
cmap=plt.cm.coolwarm, alpha=0.8)
ax.scatter(X0, X1, c=y, cmap=plt.cm.coolwarm, s=20, edgecolors='k')
ax.set_xlim(xx.min(), xx.max())
ax.set_ylim(yy.min(), yy.max())
ax.set_xlabel('Sepal length')
ax.set_ylabel('Sepal width')
ax.set_xticks(())
ax.set_yticks(())
ax.set_title(title)
plt.show()
Tiempo total de ejecución del script: (0 minutos 0.821 segundos)
