Nota
Haz clic aquí para descargar el código completo del ejemplo o para ejecutar este ejemplo en tu navegador a través de Binder
Estimación Simple de la Densidad Núcleo 1D¶
Este ejemplo utiliza la clase KernelDensity para demostrar los principios de Estimación de Densidad Núcleo en una dimensión.
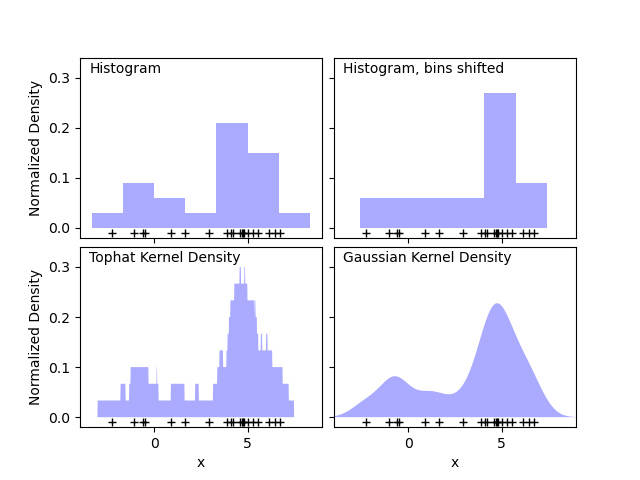
El primer gráfico muestra uno de los problemas del uso de histogramas para visualizar la densidad de puntos en 1D. Intuitivamente, un histograma puede considerarse como un esquema en el que un «bloque» unitario se coloca sobre cada punto en una cuadrícula regular. Sin embargo, como muestran los dos paneles superiores, la elección de la cuadrícula para estos bloques puede conducir a ideas deliberadamente divergentes, sobre la forma subyacente de la distribución de la densidad. Si, en cambio, centramos cada bloque en el punto que representa, obtenemos la estimación que se muestra en el panel inferior izquierdo. Se trata de una estimación de densidad núcleo con un núcleo de «sombrero de copa». Esta idea puede generalizarse a otras formas del núcleo: el panel inferior derecho de la primera figura muestra una estimación de densidad de núcleo Gaussiano sobre la misma distribución.
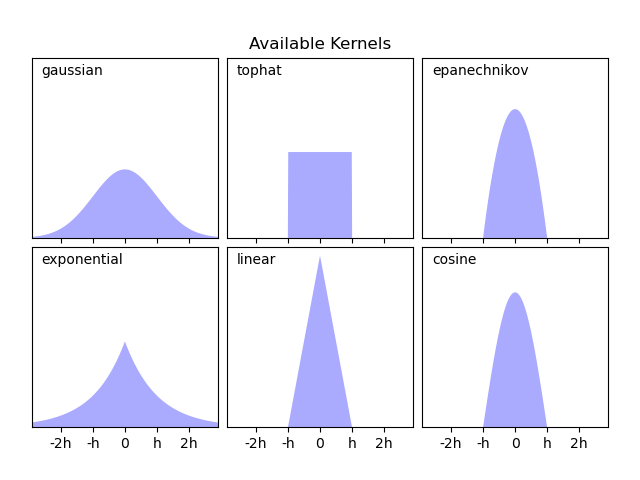
Scikit-learn implementa una estimación eficiente de la densidad del núcleo utilizando una estructura de Ball Tree o KD Tree, a través del estimador KernelDensity. Los núcleos disponibles se muestran en la segunda figura de este ejemplo.
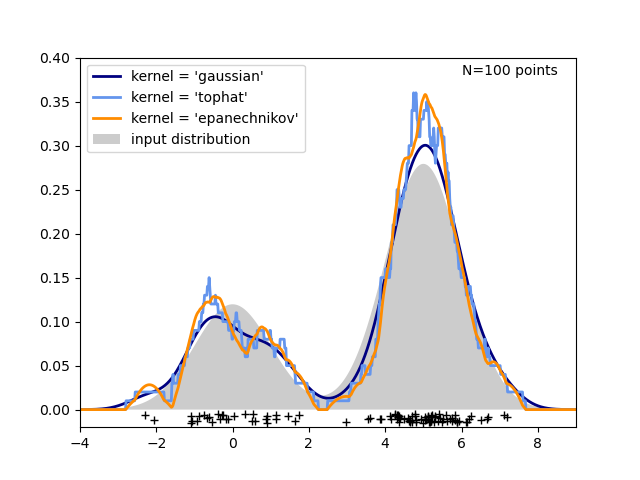
La tercera figura compara las estimaciones de la densidad núcleo para una distribución de 100 muestras en 1 dimensión. Aunque este ejemplo utiliza distribuciones 1D, la estimación de la densidad núcleo es fácil y eficazmente extensible a dimensiones más altas también.
# Author: Jake Vanderplas <jakevdp@cs.washington.edu>
#
import numpy as np
import matplotlib
import matplotlib.pyplot as plt
from scipy.stats import norm
from sklearn.neighbors import KernelDensity
from sklearn.utils.fixes import parse_version
# `normed` is being deprecated in favor of `density` in histograms
if parse_version(matplotlib.__version__) >= parse_version('2.1'):
density_param = {'density': True}
else:
density_param = {'normed': True}
# ----------------------------------------------------------------------
# Plot the progression of histograms to kernels
np.random.seed(1)
N = 20
X = np.concatenate((np.random.normal(0, 1, int(0.3 * N)),
np.random.normal(5, 1, int(0.7 * N))))[:, np.newaxis]
X_plot = np.linspace(-5, 10, 1000)[:, np.newaxis]
bins = np.linspace(-5, 10, 10)
fig, ax = plt.subplots(2, 2, sharex=True, sharey=True)
fig.subplots_adjust(hspace=0.05, wspace=0.05)
# histogram 1
ax[0, 0].hist(X[:, 0], bins=bins, fc='#AAAAFF', **density_param)
ax[0, 0].text(-3.5, 0.31, "Histogram")
# histogram 2
ax[0, 1].hist(X[:, 0], bins=bins + 0.75, fc='#AAAAFF', **density_param)
ax[0, 1].text(-3.5, 0.31, "Histogram, bins shifted")
# tophat KDE
kde = KernelDensity(kernel='tophat', bandwidth=0.75).fit(X)
log_dens = kde.score_samples(X_plot)
ax[1, 0].fill(X_plot[:, 0], np.exp(log_dens), fc='#AAAAFF')
ax[1, 0].text(-3.5, 0.31, "Tophat Kernel Density")
# Gaussian KDE
kde = KernelDensity(kernel='gaussian', bandwidth=0.75).fit(X)
log_dens = kde.score_samples(X_plot)
ax[1, 1].fill(X_plot[:, 0], np.exp(log_dens), fc='#AAAAFF')
ax[1, 1].text(-3.5, 0.31, "Gaussian Kernel Density")
for axi in ax.ravel():
axi.plot(X[:, 0], np.full(X.shape[0], -0.01), '+k')
axi.set_xlim(-4, 9)
axi.set_ylim(-0.02, 0.34)
for axi in ax[:, 0]:
axi.set_ylabel('Normalized Density')
for axi in ax[1, :]:
axi.set_xlabel('x')
# ----------------------------------------------------------------------
# Plot all available kernels
X_plot = np.linspace(-6, 6, 1000)[:, None]
X_src = np.zeros((1, 1))
fig, ax = plt.subplots(2, 3, sharex=True, sharey=True)
fig.subplots_adjust(left=0.05, right=0.95, hspace=0.05, wspace=0.05)
def format_func(x, loc):
if x == 0:
return '0'
elif x == 1:
return 'h'
elif x == -1:
return '-h'
else:
return '%ih' % x
for i, kernel in enumerate(['gaussian', 'tophat', 'epanechnikov',
'exponential', 'linear', 'cosine']):
axi = ax.ravel()[i]
log_dens = KernelDensity(kernel=kernel).fit(X_src).score_samples(X_plot)
axi.fill(X_plot[:, 0], np.exp(log_dens), '-k', fc='#AAAAFF')
axi.text(-2.6, 0.95, kernel)
axi.xaxis.set_major_formatter(plt.FuncFormatter(format_func))
axi.xaxis.set_major_locator(plt.MultipleLocator(1))
axi.yaxis.set_major_locator(plt.NullLocator())
axi.set_ylim(0, 1.05)
axi.set_xlim(-2.9, 2.9)
ax[0, 1].set_title('Available Kernels')
# ----------------------------------------------------------------------
# Plot a 1D density example
N = 100
np.random.seed(1)
X = np.concatenate((np.random.normal(0, 1, int(0.3 * N)),
np.random.normal(5, 1, int(0.7 * N))))[:, np.newaxis]
X_plot = np.linspace(-5, 10, 1000)[:, np.newaxis]
true_dens = (0.3 * norm(0, 1).pdf(X_plot[:, 0])
+ 0.7 * norm(5, 1).pdf(X_plot[:, 0]))
fig, ax = plt.subplots()
ax.fill(X_plot[:, 0], true_dens, fc='black', alpha=0.2,
label='input distribution')
colors = ['navy', 'cornflowerblue', 'darkorange']
kernels = ['gaussian', 'tophat', 'epanechnikov']
lw = 2
for color, kernel in zip(colors, kernels):
kde = KernelDensity(kernel=kernel, bandwidth=0.5).fit(X)
log_dens = kde.score_samples(X_plot)
ax.plot(X_plot[:, 0], np.exp(log_dens), color=color, lw=lw,
linestyle='-', label="kernel = '{0}'".format(kernel))
ax.text(6, 0.38, "N={0} points".format(N))
ax.legend(loc='upper left')
ax.plot(X[:, 0], -0.005 - 0.01 * np.random.random(X.shape[0]), '+k')
ax.set_xlim(-4, 9)
ax.set_ylim(-0.02, 0.4)
plt.show()
Tiempo total de ejecución del script: (0 minutos 0.514 segundos)