Nota
Haz clic aquí para descargar el código completo del ejemplo o para ejecutar este ejemplo en tu navegador a través de Binder
Visualización del comportamiento de la validación cruzada en scikit-learn¶
La elección del objeto de validación cruzada adecuado es una parte crucial para ajustar un modelo correctamente. Hay muchas formas de dividir los datos en conjuntos de entrenamiento y de prueba para evitar el sobreajuste del modelo, para estandarizar el número de grupos en los conjuntos de prueba, etc.
Este ejemplo visualiza el comportamiento de varios objetos comunes de scikit-learn para su comparación.
from sklearn.model_selection import (TimeSeriesSplit, KFold, ShuffleSplit,
StratifiedKFold, GroupShuffleSplit,
GroupKFold, StratifiedShuffleSplit)
import numpy as np
import matplotlib.pyplot as plt
from matplotlib.patches import Patch
np.random.seed(1338)
cmap_data = plt.cm.Paired
cmap_cv = plt.cm.coolwarm
n_splits = 4
Visualizar nuestros datos¶
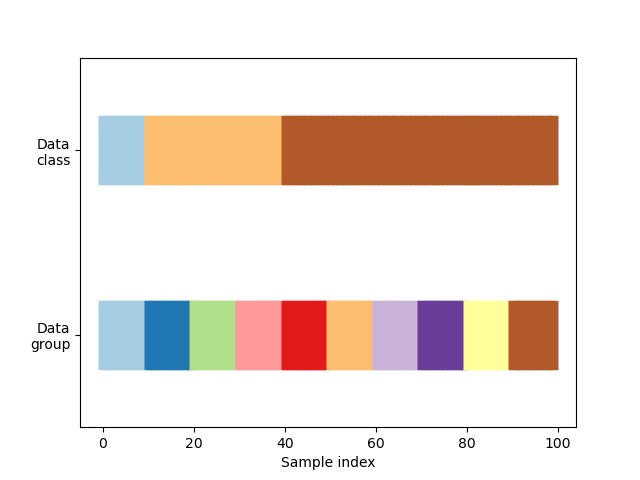
En primer lugar, debemos entender la estructura de nuestros datos. Tiene 100 puntos de datos de entrada generados aleatoriamente, 3 clases divididas de forma desigual entre los puntos de datos y 10 «grupos» divididos de forma uniforme entre los puntos de datos.
Como veremos, algunos objetos de validación cruzada hacen cosas específicas con los datos etiquetados, otros se comportan de forma diferente con los datos agrupados y otros no utilizan esta información.
Para empezar, visualizaremos nuestros datos.
# Generate the class/group data
n_points = 100
X = np.random.randn(100, 10)
percentiles_classes = [.1, .3, .6]
y = np.hstack([[ii] * int(100 * perc)
for ii, perc in enumerate(percentiles_classes)])
# Evenly spaced groups repeated once
groups = np.hstack([[ii] * 10 for ii in range(10)])
def visualize_groups(classes, groups, name):
# Visualize dataset groups
fig, ax = plt.subplots()
ax.scatter(range(len(groups)), [.5] * len(groups), c=groups, marker='_',
lw=50, cmap=cmap_data)
ax.scatter(range(len(groups)), [3.5] * len(groups), c=classes, marker='_',
lw=50, cmap=cmap_data)
ax.set(ylim=[-1, 5], yticks=[.5, 3.5],
yticklabels=['Data\ngroup', 'Data\nclass'], xlabel="Sample index")
visualize_groups(y, groups, 'no groups')
Definir una función para visualizar el comportamiento de la validación cruzada¶
Definiremos una función que nos permita visualizar el comportamiento de cada objeto de validación cruzada. Realizaremos 4 divisiones de los datos. En cada división, visualizaremos los índices elegidos para el conjunto de entrenamiento (en azul) y el conjunto de prueba (en rojo).
def plot_cv_indices(cv, X, y, group, ax, n_splits, lw=10):
"""Create a sample plot for indices of a cross-validation object."""
# Generate the training/testing visualizations for each CV split
for ii, (tr, tt) in enumerate(cv.split(X=X, y=y, groups=group)):
# Fill in indices with the training/test groups
indices = np.array([np.nan] * len(X))
indices[tt] = 1
indices[tr] = 0
# Visualize the results
ax.scatter(range(len(indices)), [ii + .5] * len(indices),
c=indices, marker='_', lw=lw, cmap=cmap_cv,
vmin=-.2, vmax=1.2)
# Plot the data classes and groups at the end
ax.scatter(range(len(X)), [ii + 1.5] * len(X),
c=y, marker='_', lw=lw, cmap=cmap_data)
ax.scatter(range(len(X)), [ii + 2.5] * len(X),
c=group, marker='_', lw=lw, cmap=cmap_data)
# Formatting
yticklabels = list(range(n_splits)) + ['class', 'group']
ax.set(yticks=np.arange(n_splits+2) + .5, yticklabels=yticklabels,
xlabel='Sample index', ylabel="CV iteration",
ylim=[n_splits+2.2, -.2], xlim=[0, 100])
ax.set_title('{}'.format(type(cv).__name__), fontsize=15)
return ax
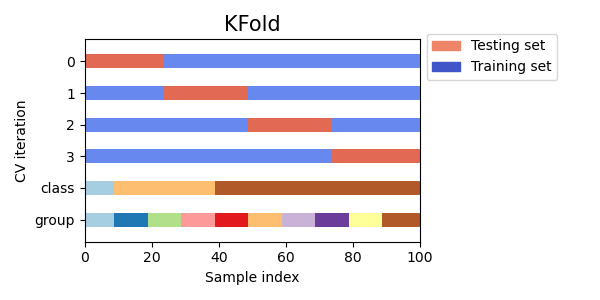
Veamos cómo se ve el objeto de validación cruzada KFold:
fig, ax = plt.subplots()
cv = KFold(n_splits)
plot_cv_indices(cv, X, y, groups, ax, n_splits)
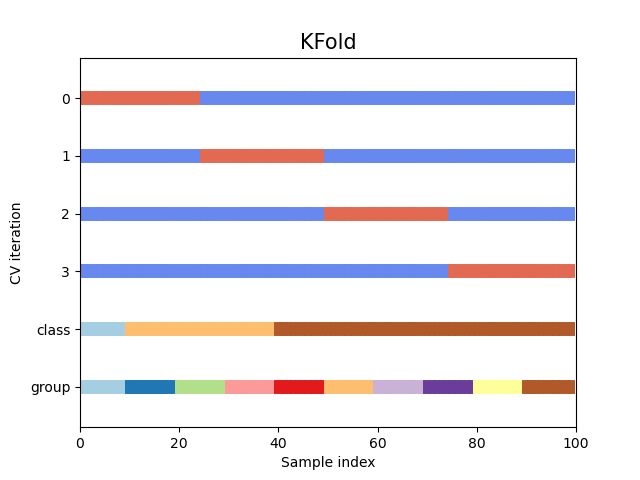
Out:
<AxesSubplot:title={'center':'KFold'}, xlabel='Sample index', ylabel='CV iteration'>
Como puedes ver, por defecto el iterador de validación cruzada KFold no tiene en cuenta ni la clase del punto de datos ni el grupo. Podemos cambiar esto utilizando el StratifiedKFold así.
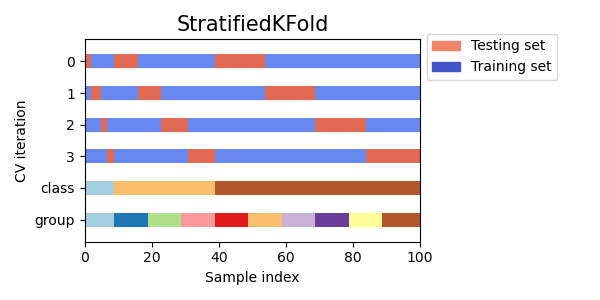
fig, ax = plt.subplots()
cv = StratifiedKFold(n_splits)
plot_cv_indices(cv, X, y, groups, ax, n_splits)
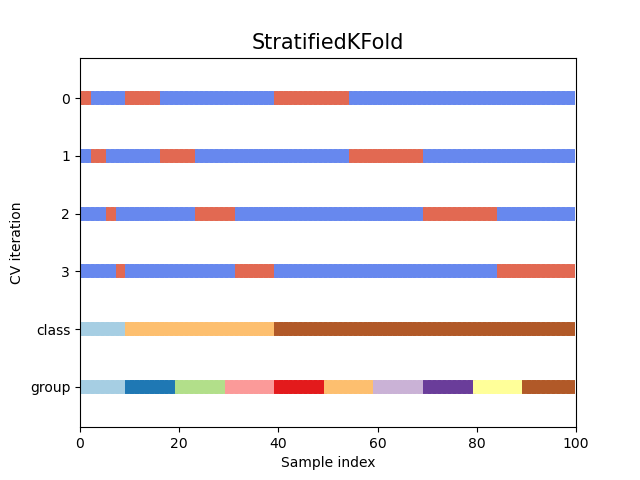
Out:
<AxesSubplot:title={'center':'StratifiedKFold'}, xlabel='Sample index', ylabel='CV iteration'>
En este caso, la validación cruzada mantuvo la misma razón(ratio) de clases en cada división de CV. A continuación, visualizaremos este comportamiento para una serie de iteradores de CV.
Visualizar los índices de validación cruzada para muchos objetos de CV¶
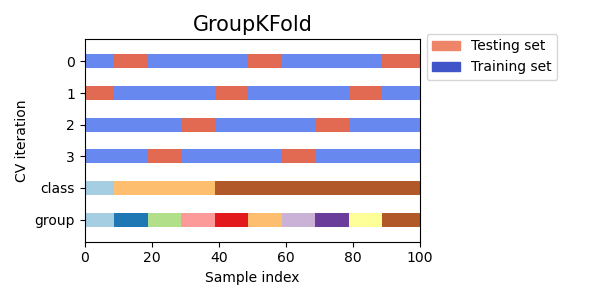
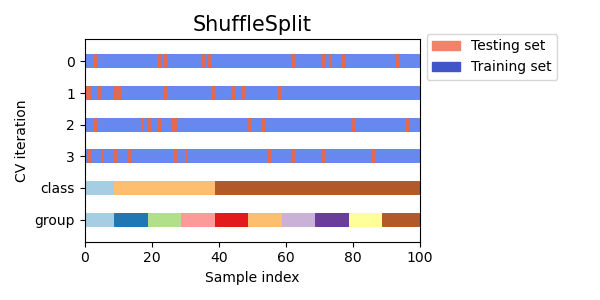
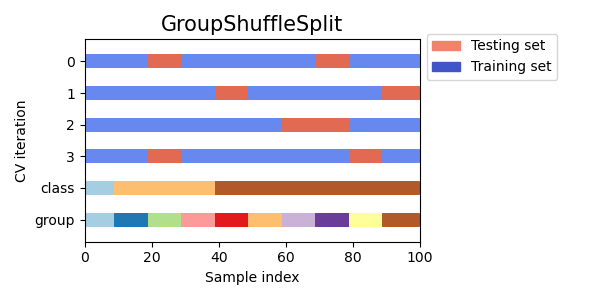
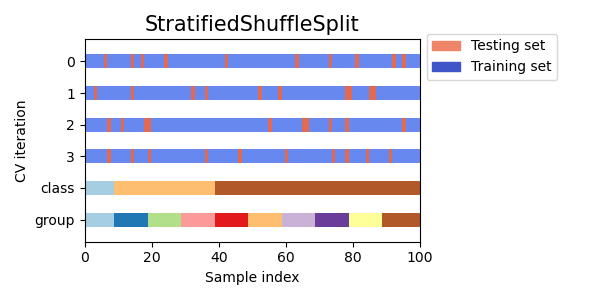
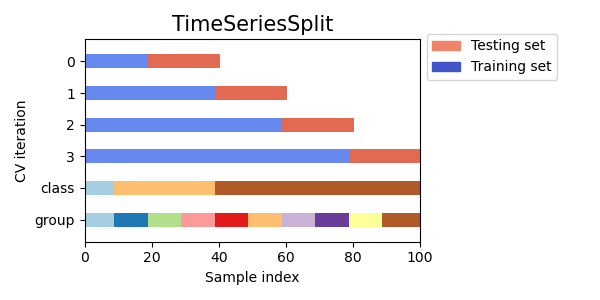
Vamos a comparar visualmente el comportamiento de la validación cruzada para muchos objetos de validación cruzada de scikit-learn. A continuación, haremos un bucle a través de varios objetos de validación cruzada comunes, visualizando el comportamiento de cada uno.
Ten en cuenta que algunos utilizan la información del grupo/clase mientras que otros no lo hacen.
cvs = [KFold, GroupKFold, ShuffleSplit, StratifiedKFold,
GroupShuffleSplit, StratifiedShuffleSplit, TimeSeriesSplit]
for cv in cvs:
this_cv = cv(n_splits=n_splits)
fig, ax = plt.subplots(figsize=(6, 3))
plot_cv_indices(this_cv, X, y, groups, ax, n_splits)
ax.legend([Patch(color=cmap_cv(.8)), Patch(color=cmap_cv(.02))],
['Testing set', 'Training set'], loc=(1.02, .8))
# Make the legend fit
plt.tight_layout()
fig.subplots_adjust(right=.7)
plt.show()
Tiempo total de ejecución del script: (0 minutos 1.446 segundos)