Nota
Haz clic aquí para descargar el código completo del ejemplo o para ejecutar este ejemplo en tu navegador a través de Binder
Aproximación explícita del mapeo de características para los núcleos RBF¶
Un ejemplo que ilustra la aproximación del mapeo de características de un núcleo RBF.
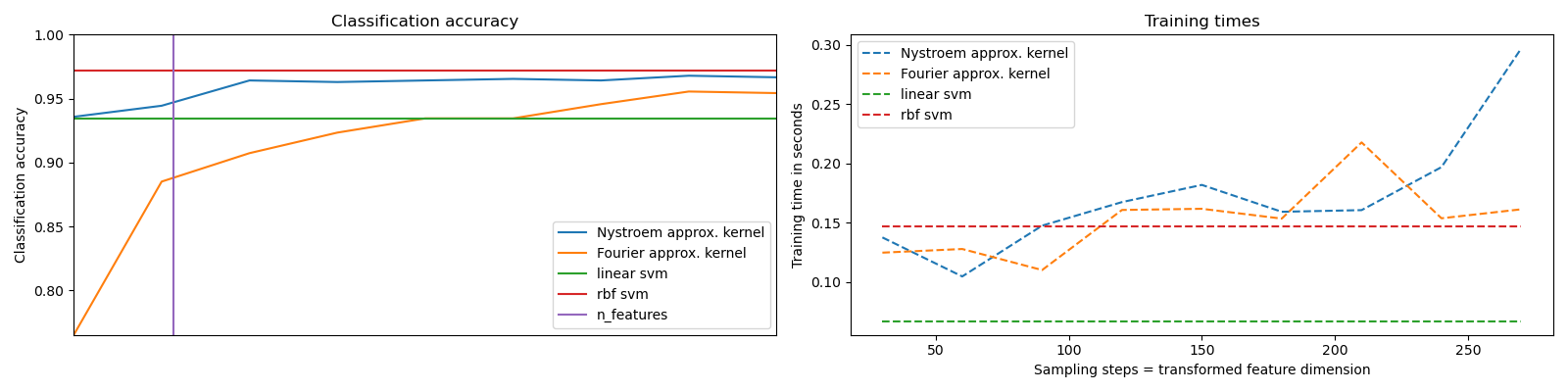
Se muestra cómo utilizar RBFSampler y Nystroem para aproximar el mapeo de características de un kernel RBF para la clasificación con una SVM en el conjunto de datos de dígitos. Se comparan los resultados utilizando una SVM lineal en el espacio original, una SVM lineal utilizando los mapeos aproximados y utilizando una SVM kernelizada. Se muestran los tiempos y la precisión para diferentes cantidades de muestreos de Monte Carlo (en el caso de RBFSampler, que utiliza características aleatorias de Fourier) y subconjuntos de diferente tamaño del conjunto de entrenamiento (para Nystroem) para el mapeo aproximado.
Ten en cuenta que el conjunto de datos aquí no es lo suficientemente grande como para mostrar los beneficios de la aproximación núcleo, ya que la SVM exacta sigue siendo razonablemente rápida.
El muestreo de más dimensiones conduce claramente a mejores resultados de clasificación, pero tiene un costo mayor. Esto significa que hay una compensación entre el tiempo de ejecución y la precisión, dada por el parámetro n_components. Ten en cuenta que la resolución de la SVM lineal y también de la SVM de aproximación núcleo podría acelerarse en gran medida mediante el uso del descenso de gradiente estocástico a través de SGDClassifier. Esto no es posible fácilmente para el caso de la SVM kernelizada.
Importación de paquetes y conjuntos de datos de Python, carga de conjuntos de datos¶
# Author: Gael Varoquaux <gael dot varoquaux at normalesup dot org>
# Andreas Mueller <amueller@ais.uni-bonn.de>
# License: BSD 3 clause
print(__doc__)
# Standard scientific Python imports
import matplotlib.pyplot as plt
import numpy as np
from time import time
# Import datasets, classifiers and performance metrics
from sklearn import datasets, svm, pipeline
from sklearn.kernel_approximation import (RBFSampler,
Nystroem)
from sklearn.decomposition import PCA
# The digits dataset
digits = datasets.load_digits(n_class=9)
Gráficos de tiempo y precisión¶
Para aplicar un clasificador sobre estos datos, necesitamos aplanar la imagen, para convertir los datos en una matriz (samples, feature):
n_samples = len(digits.data)
data = digits.data / 16.
data -= data.mean(axis=0)
# We learn the digits on the first half of the digits
data_train, targets_train = (data[:n_samples // 2],
digits.target[:n_samples // 2])
# Now predict the value of the digit on the second half:
data_test, targets_test = (data[n_samples // 2:],
digits.target[n_samples // 2:])
# data_test = scaler.transform(data_test)
# Create a classifier: a support vector classifier
kernel_svm = svm.SVC(gamma=.2)
linear_svm = svm.LinearSVC()
# create pipeline from kernel approximation
# and linear svm
feature_map_fourier = RBFSampler(gamma=.2, random_state=1)
feature_map_nystroem = Nystroem(gamma=.2, random_state=1)
fourier_approx_svm = pipeline.Pipeline([("feature_map", feature_map_fourier),
("svm", svm.LinearSVC())])
nystroem_approx_svm = pipeline.Pipeline([("feature_map", feature_map_nystroem),
("svm", svm.LinearSVC())])
# fit and predict using linear and kernel svm:
kernel_svm_time = time()
kernel_svm.fit(data_train, targets_train)
kernel_svm_score = kernel_svm.score(data_test, targets_test)
kernel_svm_time = time() - kernel_svm_time
linear_svm_time = time()
linear_svm.fit(data_train, targets_train)
linear_svm_score = linear_svm.score(data_test, targets_test)
linear_svm_time = time() - linear_svm_time
sample_sizes = 30 * np.arange(1, 10)
fourier_scores = []
nystroem_scores = []
fourier_times = []
nystroem_times = []
for D in sample_sizes:
fourier_approx_svm.set_params(feature_map__n_components=D)
nystroem_approx_svm.set_params(feature_map__n_components=D)
start = time()
nystroem_approx_svm.fit(data_train, targets_train)
nystroem_times.append(time() - start)
start = time()
fourier_approx_svm.fit(data_train, targets_train)
fourier_times.append(time() - start)
fourier_score = fourier_approx_svm.score(data_test, targets_test)
nystroem_score = nystroem_approx_svm.score(data_test, targets_test)
nystroem_scores.append(nystroem_score)
fourier_scores.append(fourier_score)
# plot the results:
plt.figure(figsize=(16, 4))
accuracy = plt.subplot(121)
# second y axis for timings
timescale = plt.subplot(122)
accuracy.plot(sample_sizes, nystroem_scores, label="Nystroem approx. kernel")
timescale.plot(sample_sizes, nystroem_times, '--',
label='Nystroem approx. kernel')
accuracy.plot(sample_sizes, fourier_scores, label="Fourier approx. kernel")
timescale.plot(sample_sizes, fourier_times, '--',
label='Fourier approx. kernel')
# horizontal lines for exact rbf and linear kernels:
accuracy.plot([sample_sizes[0], sample_sizes[-1]],
[linear_svm_score, linear_svm_score], label="linear svm")
timescale.plot([sample_sizes[0], sample_sizes[-1]],
[linear_svm_time, linear_svm_time], '--', label='linear svm')
accuracy.plot([sample_sizes[0], sample_sizes[-1]],
[kernel_svm_score, kernel_svm_score], label="rbf svm")
timescale.plot([sample_sizes[0], sample_sizes[-1]],
[kernel_svm_time, kernel_svm_time], '--', label='rbf svm')
# vertical line for dataset dimensionality = 64
accuracy.plot([64, 64], [0.7, 1], label="n_features")
# legends and labels
accuracy.set_title("Classification accuracy")
timescale.set_title("Training times")
accuracy.set_xlim(sample_sizes[0], sample_sizes[-1])
accuracy.set_xticks(())
accuracy.set_ylim(np.min(fourier_scores), 1)
timescale.set_xlabel("Sampling steps = transformed feature dimension")
accuracy.set_ylabel("Classification accuracy")
timescale.set_ylabel("Training time in seconds")
accuracy.legend(loc='best')
timescale.legend(loc='best')
plt.tight_layout()
plt.show()
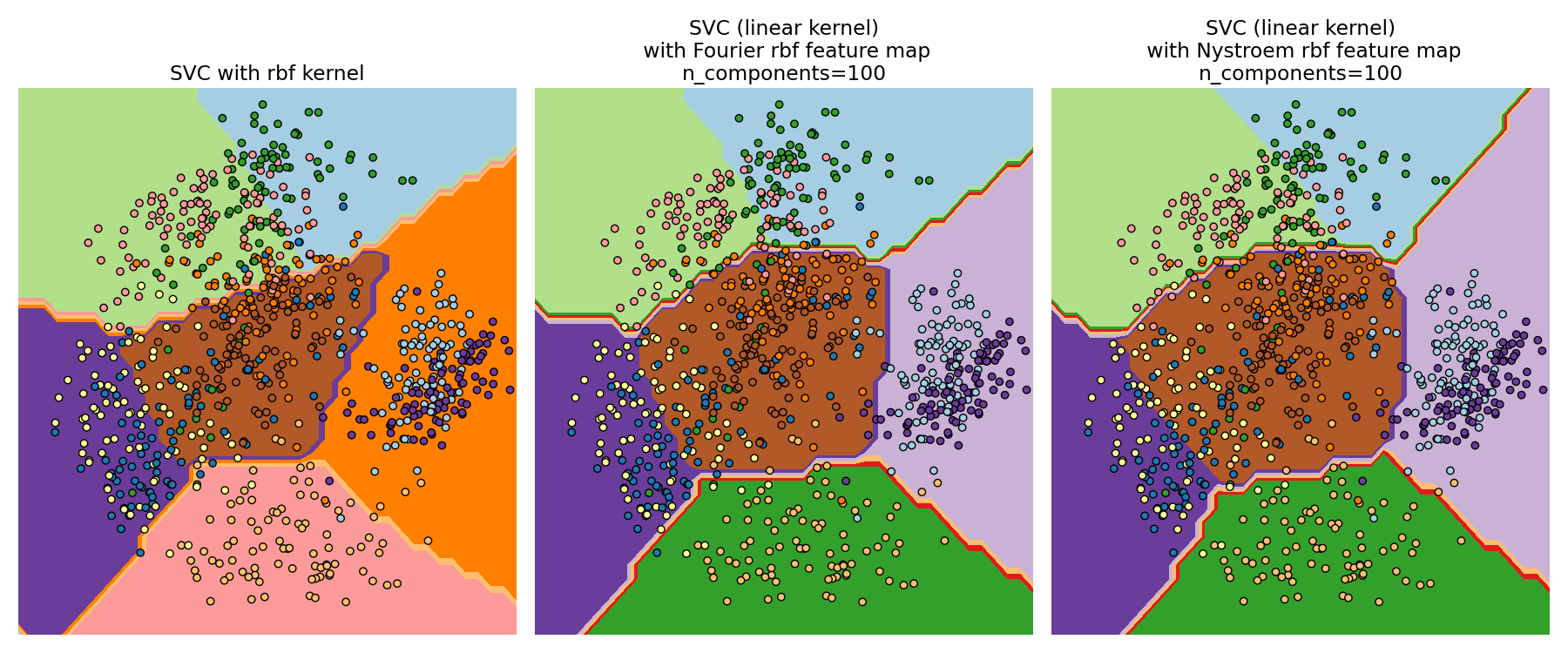
Superficies de decisión de la SVM de núcleo RBF y la SVM lineal¶
El segundo gráfico visualiza las superficies de decisión de la SVM de núcleo RBF y la SVM lineal con mapeo de aproximación núcleo. El gráfico muestra las superficies de decisión de los clasificadores proyectadas sobre los dos primeros componentes principales de los datos. Esta visualización debe ser tomada con cautela ya que es sólo un corte interesante a través de la superficie de decisión en 64 dimensiones. En particular, ten en cuenta que un punto de datos (representado como un punto) no se clasifica necesariamente en la región en la que se encuentra, ya que no se ubicará en el plano que abarcan los dos primeros componentes principales. El uso de RBFSampler y Nystroem se describe en detalle en Aproximación de núcleo.
# visualize the decision surface, projected down to the first
# two principal components of the dataset
pca = PCA(n_components=8).fit(data_train)
X = pca.transform(data_train)
# Generate grid along first two principal components
multiples = np.arange(-2, 2, 0.1)
# steps along first component
first = multiples[:, np.newaxis] * pca.components_[0, :]
# steps along second component
second = multiples[:, np.newaxis] * pca.components_[1, :]
# combine
grid = first[np.newaxis, :, :] + second[:, np.newaxis, :]
flat_grid = grid.reshape(-1, data.shape[1])
# title for the plots
titles = ['SVC with rbf kernel',
'SVC (linear kernel)\n with Fourier rbf feature map\n'
'n_components=100',
'SVC (linear kernel)\n with Nystroem rbf feature map\n'
'n_components=100']
plt.figure(figsize=(18, 7.5))
plt.rcParams.update({'font.size': 14})
# predict and plot
for i, clf in enumerate((kernel_svm, nystroem_approx_svm,
fourier_approx_svm)):
# Plot the decision boundary. For that, we will assign a color to each
# point in the mesh [x_min, x_max]x[y_min, y_max].
plt.subplot(1, 3, i + 1)
Z = clf.predict(flat_grid)
# Put the result into a color plot
Z = Z.reshape(grid.shape[:-1])
plt.contourf(multiples, multiples, Z, cmap=plt.cm.Paired)
plt.axis('off')
# Plot also the training points
plt.scatter(X[:, 0], X[:, 1], c=targets_train, cmap=plt.cm.Paired,
edgecolors=(0, 0, 0))
plt.title(titles[i])
plt.tight_layout()
plt.show()
Tiempo total de ejecución del script: (0 minutos 5.556 segundos)
