Nota
Haz clic en aquí para descargar el código de ejemplo completo o para ejecutar este ejemplo en tu navegador a través de Binder
Ajuste del estimador lineal robusto¶
Aquí se ajusta una función seno con un polinomio de orden 3, para valores cercanos a cero.
El ajuste robusto se demuestra en diferentes situaciones:
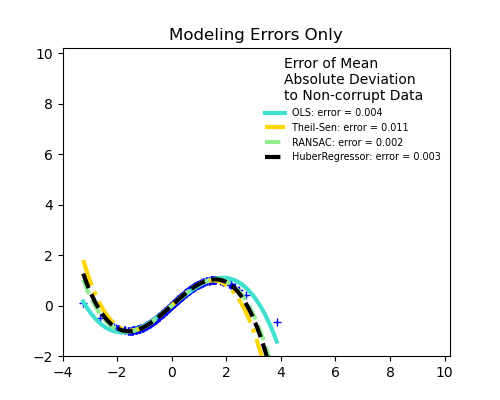
No hay errores de medición, sólo de modelización (ajuste de un seno con un polinomio)
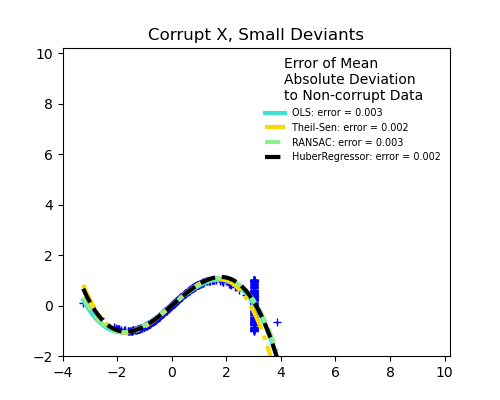
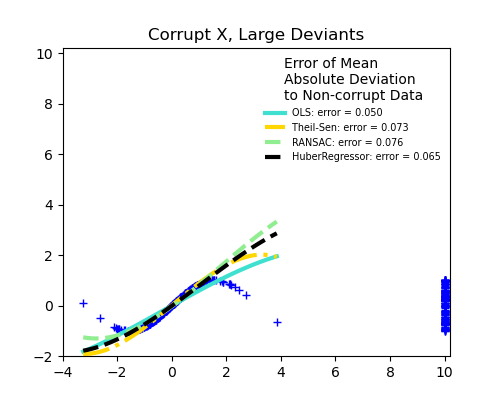
Errores de medición en X
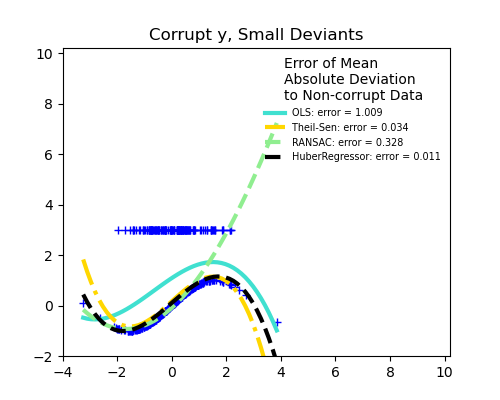
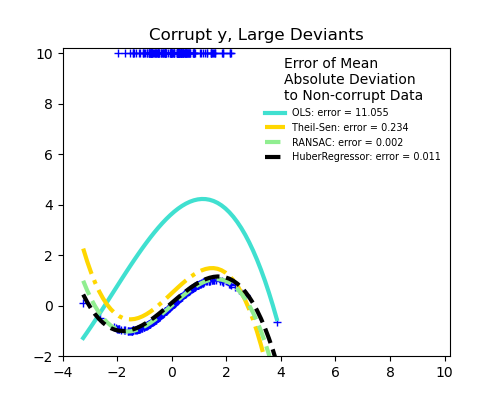
Errores de medición en y
La mediana de la desviación absoluta respecto a los nuevos datos no corruptos se utiliza para juzgar la calidad de la predicción.
Lo que podemos ver que:
RANSAC es bueno para los valores atípicos fuertes en la dirección y
TheilSen es bueno para los valores atípicos pequeños, tanto en la dirección X como en la y, pero tiene un punto de ruptura por encima del cual funciona peor que OLS.
Las puntuaciones de HuberRegressor no pueden compararse directamente con las de TheilSen y RANSAC porque no intenta filtrar completamente los valores atípicos, sino disminuir su efecto.
from matplotlib import pyplot as plt
import numpy as np
from sklearn.linear_model import (
LinearRegression, TheilSenRegressor, RANSACRegressor, HuberRegressor)
from sklearn.metrics import mean_squared_error
from sklearn.preprocessing import PolynomialFeatures
from sklearn.pipeline import make_pipeline
np.random.seed(42)
X = np.random.normal(size=400)
y = np.sin(X)
# Make sure that it X is 2D
X = X[:, np.newaxis]
X_test = np.random.normal(size=200)
y_test = np.sin(X_test)
X_test = X_test[:, np.newaxis]
y_errors = y.copy()
y_errors[::3] = 3
X_errors = X.copy()
X_errors[::3] = 3
y_errors_large = y.copy()
y_errors_large[::3] = 10
X_errors_large = X.copy()
X_errors_large[::3] = 10
estimators = [('OLS', LinearRegression()),
('Theil-Sen', TheilSenRegressor(random_state=42)),
('RANSAC', RANSACRegressor(random_state=42)),
('HuberRegressor', HuberRegressor())]
colors = {'OLS': 'turquoise', 'Theil-Sen': 'gold', 'RANSAC': 'lightgreen', 'HuberRegressor': 'black'}
linestyle = {'OLS': '-', 'Theil-Sen': '-.', 'RANSAC': '--', 'HuberRegressor': '--'}
lw = 3
x_plot = np.linspace(X.min(), X.max())
for title, this_X, this_y in [
('Modeling Errors Only', X, y),
('Corrupt X, Small Deviants', X_errors, y),
('Corrupt y, Small Deviants', X, y_errors),
('Corrupt X, Large Deviants', X_errors_large, y),
('Corrupt y, Large Deviants', X, y_errors_large)]:
plt.figure(figsize=(5, 4))
plt.plot(this_X[:, 0], this_y, 'b+')
for name, estimator in estimators:
model = make_pipeline(PolynomialFeatures(3), estimator)
model.fit(this_X, this_y)
mse = mean_squared_error(model.predict(X_test), y_test)
y_plot = model.predict(x_plot[:, np.newaxis])
plt.plot(x_plot, y_plot, color=colors[name], linestyle=linestyle[name],
linewidth=lw, label='%s: error = %.3f' % (name, mse))
legend_title = 'Error of Mean\nAbsolute Deviation\nto Non-corrupt Data'
legend = plt.legend(loc='upper right', frameon=False, title=legend_title,
prop=dict(size='x-small'))
plt.xlim(-4, 10.2)
plt.ylim(-2, 10.2)
plt.title(title)
plt.show()
Tiempo total de ejecución del script: (0 minutos 3.470 segundos)