Nota
Haz clic en aquí para descargar el código de ejemplo completo o para ejecutar este ejemplo en tu navegador a través de Binder
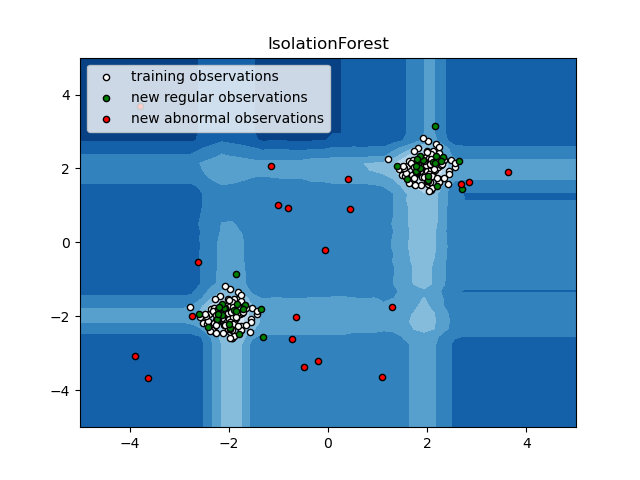
Ejemplo de IsolationForest¶
Un ejemplo utilizando IsolationForest para la detección de anomalías.
El IsolationForest «aísla» las observaciones seleccionando aleatoriamente una característica y luego seleccionando aleatoriamente un valor de división entre los valores máximos y mínimos de la característica seleccionada.
Dado que la partición recursiva puede representarse mediante una estructura de árbol, el número de particiones necesarias para aislar una muestra es equivalente a la longitud del camino desde el nodo raíz hasta el nodo final.
Esta longitud del recorrido, promediada sobre un bosque de tales árboles aleatorios, es una medida de normalidad y nuestra función de decisión.
La partición aleatoria produce trayectorias notablemente más cortas para las anomalías. Por lo tanto, cuando un bosque de árboles aleatorios produce colectivamente trayectorias más cortas para determinadas muestras, es muy probable que se trate de anomalías.
print(__doc__)
import numpy as np
import matplotlib.pyplot as plt
from sklearn.ensemble import IsolationForest
rng = np.random.RandomState(42)
# Generate train data
X = 0.3 * rng.randn(100, 2)
X_train = np.r_[X + 2, X - 2]
# Generate some regular novel observations
X = 0.3 * rng.randn(20, 2)
X_test = np.r_[X + 2, X - 2]
# Generate some abnormal novel observations
X_outliers = rng.uniform(low=-4, high=4, size=(20, 2))
# fit the model
clf = IsolationForest(max_samples=100, random_state=rng)
clf.fit(X_train)
y_pred_train = clf.predict(X_train)
y_pred_test = clf.predict(X_test)
y_pred_outliers = clf.predict(X_outliers)
# plot the line, the samples, and the nearest vectors to the plane
xx, yy = np.meshgrid(np.linspace(-5, 5, 50), np.linspace(-5, 5, 50))
Z = clf.decision_function(np.c_[xx.ravel(), yy.ravel()])
Z = Z.reshape(xx.shape)
plt.title("IsolationForest")
plt.contourf(xx, yy, Z, cmap=plt.cm.Blues_r)
b1 = plt.scatter(X_train[:, 0], X_train[:, 1], c='white',
s=20, edgecolor='k')
b2 = plt.scatter(X_test[:, 0], X_test[:, 1], c='green',
s=20, edgecolor='k')
c = plt.scatter(X_outliers[:, 0], X_outliers[:, 1], c='red',
s=20, edgecolor='k')
plt.axis('tight')
plt.xlim((-5, 5))
plt.ylim((-5, 5))
plt.legend([b1, b2, c],
["training observations",
"new regular observations", "new abnormal observations"],
loc="upper left")
plt.show()
Tiempo total de ejecución del script: (0 minutos 0.591 segundos)
